来源:科研圈
细胞分裂过程中,遗传物质 DNA 的均等分配至关重要,而凝缩蛋白有着关键作用,科学家们一直在探索凝缩蛋白的具体作用机制。近日,科学家们发现了 DNA 一种全新的折叠方式——“Z 型环”,为更好地了解 DNA 迈出了重要的一步。

图片来源:Pixabay
来源:代尔夫特理工大学
翻译:阿金
审校/编辑:李光昭
细胞中的 DNA 形态堪比餐盘中的意大利面条:杂乱地纠缠成团。为了在细胞分裂时,能够将遗传物质 DNA 均等地分给两个子细胞,细胞将这团“乱麻”浓缩成染色体的形式。在这一过程中,一种蛋白复合物——凝缩蛋白(condensin)发挥了关键作用,但生物学家还不清楚其确切的分子机制。直到 2018 年 2 月,代尔夫特理工大学(Delft University of Technology)卡夫里研究所(Kavli Institute)的科学家们和海德堡欧洲分子生物学实验室(EMBL Heidelberg)的同事们一起通过实时成像技术,展示了凝缩蛋白如何将 DNA 挤压成环状结构。如今,来自同一研究小组的后续研究表明,这绝不是凝缩蛋白凝聚 DNA 的唯一方式。研究人员发现了一种全新的 DNA 环状结构,并将其称为“Z 型环”(Z-loop)。他们将这一新现象发表在 3 月 4 日的《自然》(Nature)杂志上,首次揭示了凝缩蛋白如何通过相互作用,将 DNA 折叠形成 Z 型结构。
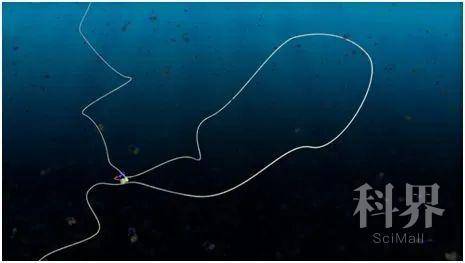
图片来源:Cees Dekker Lab TU Delft/Scixel
环状结构以外
代尔夫特理工大学的博士后,欧根·金博士(Dr. Eugene Kim)说:“这一研究源起自一个疑问:DNA 是否可以仅通过环状结构的折叠浓缩成染色体,还是有其它我们不了解的环节。我们希望可以同时看到几个凝缩蛋白。在实验过程中,我们发现了一种全新的 DNA 折叠形式,和单环结构有着显著差别,而且令人意外的是,比单环结构的出现比例更高。我们通过实验手段确认,DNA 被折叠成了一种“之字形”结构。因为 DNA 折叠后的形状像英文字母 Z,我们将这种结构命名为‘Z 型环’。研究人员们因为好奇开始研究这一结构。“这很出乎我们的意料。”金博士继续讲到,“我们在猜想,DNA 如何通过两个凝缩蛋白形成这样的结构?这其中的分子机制是什么?”
合作完成的Z型结构
研究领头人塞斯·德克教授(Cees Dekker)解释道:“Z 型结构的形成,先由一个凝缩蛋白着陆在 DNA 表面,产生单个 DNA 环开始,随后,第二个凝缩蛋白结合 DNA 单环,开始制造第二个单环,形成环中环。当两个凝缩蛋白像拔河一样向两边拉锯时,出现了一个令人惊讶的现象了:第二个凝缩蛋白跳过了第一个凝缩蛋白,捕获环外的 DNA,并沿着 DNA 继续运动。我们感到很吃惊:两个凝缩蛋白复合物竟然可以穿过彼此。这与现有模型并不相符:科学家曾认为凝缩蛋白相遇时,会相互阻碍。”

图片来源:DNA-loop extruding condensin complexes can traverse one another,Eugene Kim et al.
观察工作中的凝缩蛋白
在细胞中 DNA 的缠绕方式非常复杂,因此很难将其分离出来,研究成环的过程。因此,研究人员决定在玻璃片上观察单个 DNA 分子的成环过程。他们将 DNA 分子的两端固定在玻璃片表面,并通过荧光标记 DNA 分子和凝缩蛋白。然后,垂直于分子方向,向液体施加流动,研究人员成功使 DNA 呈现 U 型,放到显微镜下进行观察。
医学研究意义
该研究为我们从根本上了解细胞中的 DNA 迈出了重要的一步。同时,还具有医学研究意义。凝缩蛋白所属的染色体结构维持(SMC)蛋白家族的异常与多种遗传性疾病相关,例如德朗热综合症 (CdLS)。凝缩蛋白对细胞分裂过程中的染色体分配和变化也发挥着关键作用,而染色体(数目)变化异常,便可能导致癌变。更好地理解这其中潜在的分子机制对于研究这些严重疾病的分子起源至关重要。
原文链接:
https://www.eurekalert.org/pub_releases/2020-03/duot-zd030220.php
论文信息【标题】
DNA-loop extruding condensin complexes can traverse one another
【作者】Eugene Kim et al.
【时间】04 March 2020
【链接】
https://www.nature.com/articles/s41586-020-2067-5
【摘要】
Condensin, a key component of the structure maintenance of chromosome (SMC) protein complexes, has recently been shown to be a motor that extrudes loops of DNA1. It remains unclear, however, how condensin complexes work together to collectively package DNA into chromosomes. Here we use time-lapse single-molecule visualization to study mutual interactions between two DNA-loop-extruding yeast condensins. We find that these motor proteins, which, individually, extrude DNA in one direction only are able to dynamically change each other’s DNA loop sizes, even when far apart. When they are in close proximity, condensin complexes are able to traverse each other and form a loop structure, which we term a Z-loop—three double-stranded DNA helices aligned in parallel with one condensin at each edge. Z-loops can fill gaps left by single loops and can form symmetric dimer motors that pull in DNA from both sides. These findings indicate that condensin may achieve chromosomal compaction using a variety of looping structures.
来源:keyanquan 科研圈
原文链接:https://mp.weixin.qq.com/s?__biz=MzA5NDkzNjIwMg==&mid=2651690802&idx=3&sn=e800708f0d92cdd7432acc1c1e171aff&chksm=8bbe3ad7bcc9b3c1c494f1ef85be700a7c03e728d91ea2087e1dc22f35c3f56ea53d51ff1ecd#rd
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn


