来源:BioArt植物
原标题:PNAS: 完善promoter escape的分子机制,张余研究组捕获细菌RNA聚合酶转录起始后期的动态过程
2020年3月3日,PNAS 在线发表了中国科学院分子植物科学卓越创新中心合成生物学重点实验室张余研究组与美国Rutgers University的Richard Ebright研究组合作完成的题为“ RNA extension drives a stepwise displacement of an initiation-factor structural module in initial transcription”的研究论文。该研究捕获细菌RNA聚合酶转录起始后期的动态过程。

基因转录起始需要RNA聚合酶和转录起始因子σ组成复合物,再一起锚定到基因的启动子区域,解开双链DNA,起始RNA合成。在这个过程中,RNA聚合酶与大约60-bp DNA建立牢固的相互作用,以确保转录起始的高效进行。然而,RNAP随后必须完全挣脱上述相互作用才能启航完成RNA的延伸,这一过程称为promoter escape。RNA聚合酶从哪里获得能量,并且如何积累能量来实现promoter escape,一直是大家感兴趣的问题。
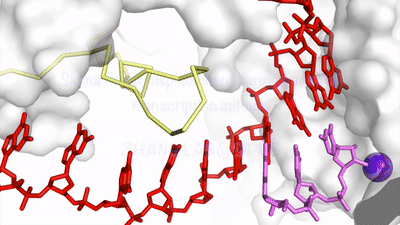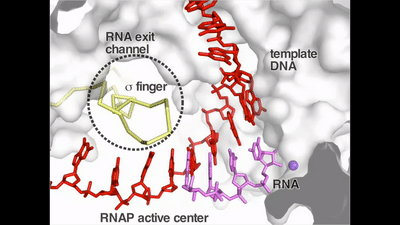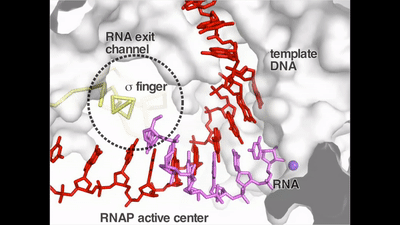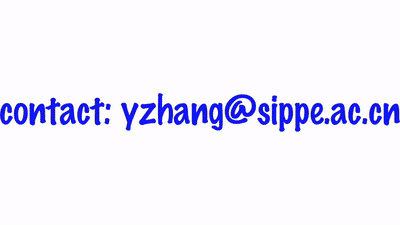
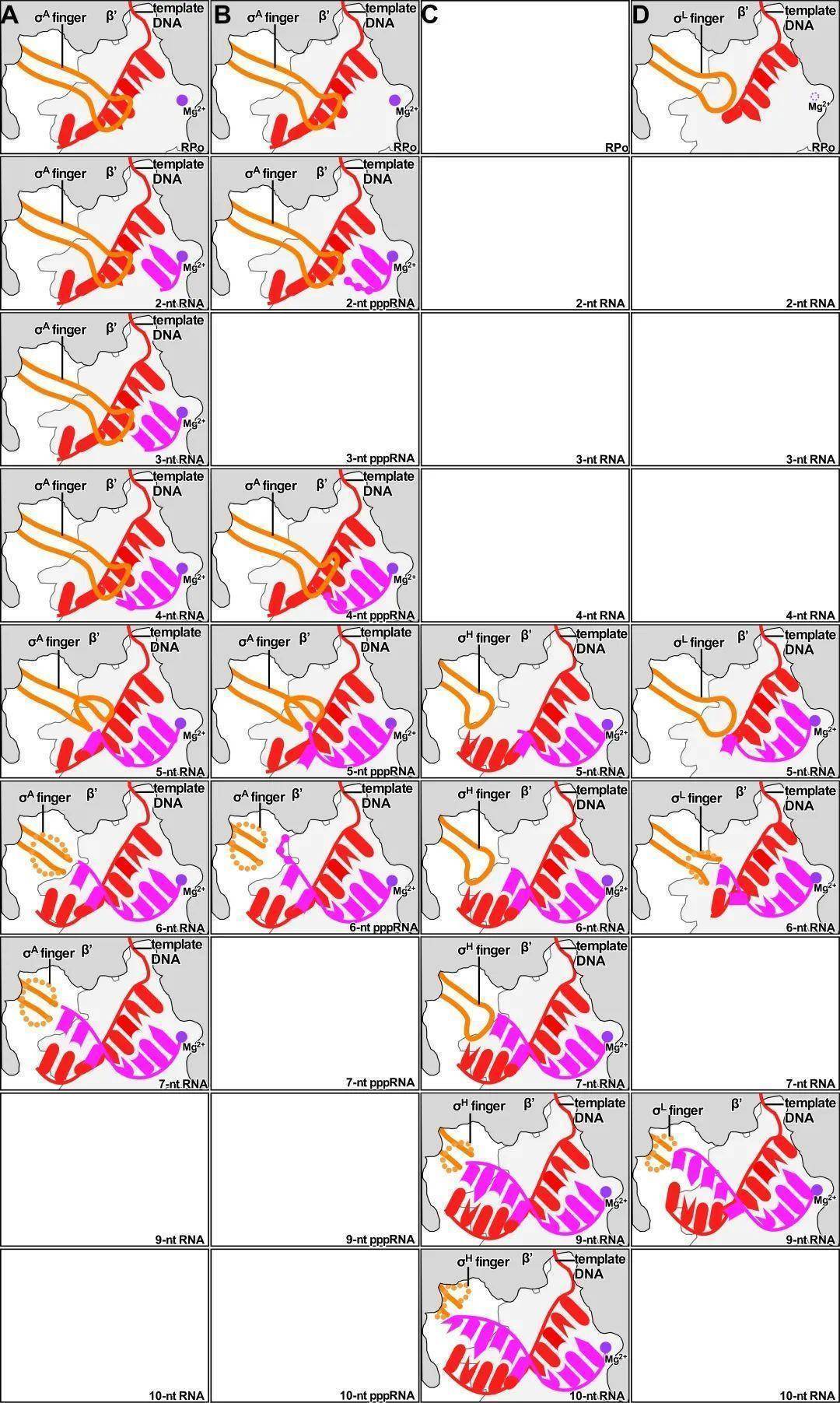
该研究发现在细菌,真核和古菌这三界生物中,转录起始因子都存在一个结构模块 (在细菌中为σ finger),这个模块深入到RNAP的催化中心,在RNA聚合酶解链双链DNA的时候,其能够稳定单链的转录泡结构。但是该结构模块的位置却堵住了新生RNA的延伸路径。在RNA的延长过程中,它势必会与RNA发生冲突。在这项研究中,作者解析了细菌转录起始阶段的大约20个复合物晶体结构,还原了这一个冲突的细节。作者发现,RNA的延长会逐渐挤压σ finger,并最终将其推出RNAP聚合酶中心,根据结构信息,作者提出了promoter escape分子机制的假设。作者认为σ finger相当于一个弹簧,RNA在刚开始延长的过程中不断的挤压这一弹簧,能够将NTP水解的自由能不断转换成机械能,并积累到σ finger的蛋白弹簧上,当这一弹簧被挤压到极限时能够促发RNAP与启动子DNA的解离,完成Promoter escape。这一假设与15年前提出的DNA弹簧(DNA scrunching)理论相互补充,完善了promoter escape的的分子机制。
张余研究组的李玲婷博士生和Rutgers university的Vadim Molodtsov博士是论文的共同第一作者,张余研究员与Rutgers university的Richard Ebright教授是该论文的共同通讯作者。
论文链接:
https://www.pnas.org/content/early/2020/03/02/1920747117
来源:bioartplants BioArt植物
原文链接:http://mp.weixin.qq.com/s?__biz=MzU3ODY3MDM0NA==&mid=2247494267&idx=4&sn=2b53c53fab02158713813d0d27726cfe&chksm=fd73701cca04f90a3d46cdc82c727d880f1e6fbaf486bc5231e421d215695b394a5f3f0f60a9&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn



