来源:中国生物物理学会
增强子是基因组中调控基因时空特异性表达的重要元件,在细胞分化、疾病发生等过程中起重要作用【1】。但是,如何从卷帙浩繁的基因组序列中精确地鉴定增强子区域和序列,依然是目前研究的焦点。真核细胞的基因组DNA在细胞内紧紧缠绕组蛋白八聚体,以染色质的形式被压缩存储、保护和行使功能。染色质上不同区域的组成特征和各类化学修饰则作为表观遗传标记,构成了解读基因组序列、寻找功能元件的信标。美国加州大学圣迭戈分校任冰实验室利用染色质免疫沉淀技术(ChIP)分析了多种组蛋白修饰在基因组上的分布,最早将特定的组蛋白修饰与启动子和增强子等重要的功能元件联系起来【2,3】。随后,MIT的Rudolf Jaenisch实验室发现利用H3K27乙酰化修饰(H3K27ac)可以有效地区分活跃增强子和非活跃的增强子【4】。除此之外,增强子还高度富集组蛋白变体H3.3,并具有较高的H3.3更新替换活性【5,6】。虽然H3K27ac指示活跃增强子的能力无可辩驳,但该修饰是否会功能性地影响增强子活性一直是领域内存有争议的问题。
2020年2月21日,中国科学院生物物理研究所朱冰课题组在Genome Biology上在线发表了题为Histone H3K27 acetylation is dispensable for enhancer activity in mouse embryonic stem cells的研究论文。该工作首次证明了虽然H3K27ac可以作为哺乳动物基因组中活跃增强子的最好的表观遗传标记,但活跃增强子的生物学功能并不受缺失H3K27ac的影响,而是依赖于组蛋白多个赖氨酸位点乙酰化的协同作用。

为了特异性地研究增强子区域H3K27ac的生物学功能,研究人员在小鼠胚胎干细胞中定点突变了组蛋白变体H3.3的第27位赖氨酸,将其突变为不能被乙酰化修饰的精氨酸(H3.3K27R)。全基因组学分析发现,在不影响H3K27位点另一个重要修饰H3K27me3的前提下,H3.3K27R突变的细胞中活跃增强子区域的H3K27ac基本完全消失,但细胞转录组几乎没有受到影响。最近开发的“Activity-by-Contact”模型可以相对较好地预测增强子与其所调控的基因之间的对应关系【7】。利用这一模型,研究人员发现基因的转录对于与其关联的增强子区域H3K27ac的消失并不敏感,仅少数基因呈现上调或者下调表达的现象。即便是对于具有高度组织特异性、且高度富集H3K27ac修饰的超级增强子(super enhancer)而言,H3.3K27R突变的细胞中超级增强子调控的基因转录也没有发生显著变化。
乙酰化修饰能够中和赖氨酸的正电性,从而削弱组蛋白与DNA结合的能力【8】。组蛋白上有多个可以被乙酰化修饰的赖氨酸位点。研究人员分析了H3.3K27R突变对组蛋白其他位点乙酰化修饰的影响,结果发现其他位点乙酰化并无明显变化,这说明增强子的活性并不依赖于单一特定位点的乙酰化修饰,而是需要多个位点乙酰化修饰协同作用,使染色质结构变得松散,从而维持增强子的活跃状态。
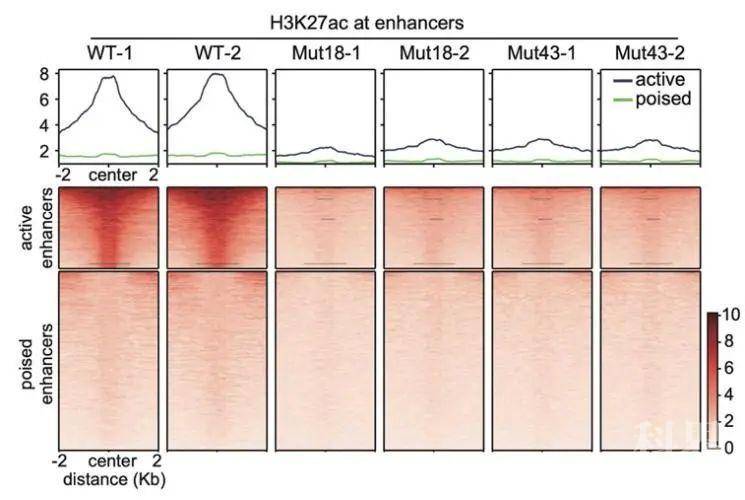
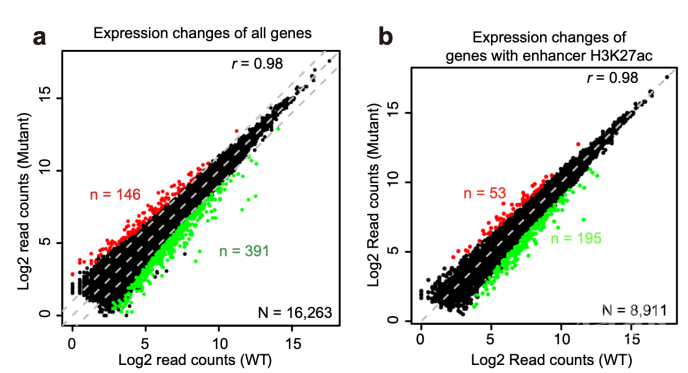
增强子区域H3K27ac的丢失不影响整体基因转录
据悉,中国科学院生物物理研究所朱冰课题组博士生张甜甜和研究员张珠强是该论文的共同第一作者,研究员朱冰和助理研究员熊俊(青促会会员)为该论文的通讯作者。朱冰课题组的董强博士也参与了这项工作。
后记:据了解,该成果的起源颇有意思。据悉,朱冰老师在一次审稿的过程中,把今日报道中的文章中的道理进行了提及,对手头审稿的文章提出了一些观点和批评,然后其它审稿人似乎并没有重视(也许不懂)而忽视了问题的存在,那个文章最后也发表了。想必朱冰老师也是忿忿不平,于是决定要把这个问题仔细做一做,让一个运气稍差但是面临毕业的学生去做了一下,于是有了这篇GB的文章。这篇GB的文章不复杂,对于很多业内的人来说这些结论早就是已知的(比如蓝斐老师昨日也说这个结论是显而易见,并且对于这个GB的工作还是很赞赏的),只是没有人去专门讲,但是对于圈外的人这个问题还是很重要的,或许有的人还会大吃一惊。总之,从某种程度上讲,这个工作的教育意义大于科学意义,个中滋味,读者不妨细细品味。引用朱冰老师的依据原话,“In science, correlation does not necessarily mean causality”。
原文链接:
https://genomebiology.biomedcentral.com/articles/10.1186/s13059-020-01957-w
参考文献
1. Calo E, Wysocka J. Modification of enhancer chromatin: what, how, and why? Mol Cell. 2013;49:825-37.
2. Heintzman ND, Stuart RK, Hon G, Fu Y, Ching CW, Hawkins RD et al. Distinct and predictive chromatin signatures of transcriptional promoters and enhancers in the human genome. Nat Genet. 2007;39:311-8.
3. Heintzman ND, Hon GC, Hawkins RD, Kheradpour P, Stark A, Harp LF et al. Histone modifications at human enhancers reflect global cell-type-specific gene expression. Nature. 2009;459:108-12.
4. Creyghton MP, Cheng AW, Welstead GG, Kooistra T, Carey BW, Steine EJ et al. Histone H3K27ac separates active from poised enhancers and predicts developmental state. Proc Natl Acad Sci USA. 2010;107:21931-6.
5. Kraushaar DC, Jin W, Maunakea A, Abraham B, Ha M, Zhao K. Genome-wide incorporation dynamics reveal distinct categories of turnover for the histone variant H3.3. Genome Biol. 2013;14:R121.
6. Deaton AM, Gomez-Rodriguez M, Mieczkowski J, Tolstorukov MY, Kundu S, Sadreyev RI et al. Enhancer regions show high histone H3.3 turnover that changes during differentiation. Elife. 2016;5.
7. Fulco CP, Nasser J, Jones TR, Munson G, Bergman DT, Subramanian V et al. Activity-by-contact model of enhancer-promoter regulation from thousands of CRISPR perturbations. Nat Genet. 2019;51:1664-9.
8. Eberharter A, Becker PB. Histone acetylation: a switch between repressive and permissive chromatin. Second in review series on chromatin dynamics. EMBO Rep. 2002;3:224-9.
本文转载自公众号“BioArt”(BioGossip)
中国生物物理学会官方订阅号,为BSC会员及生物物理领域专业人士服务。
投稿及授权请联系:bscoffice@bsc.org.cn。
微信号:BSC-1979

来源:BSC-1979 中国生物物理学会
原文链接:http://mp.weixin.qq.com/s?__biz=MzA4NjE1NTQ5Mg==&mid=2649499197&idx=2&sn=9eab930fef1a0fad37cf6524709093bd&chksm=87d58fb6b0a206a07e1022e70d7d179dfd3297eae4d455dea4fa54f780bc14a8f92e3f393e34&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn



