CRISPR/Cas9系统已成为基因组精准修饰的有效工具,有效促进了水稻功能基因组学研究和分子育种进程。然而Cas9蛋白需识别特定的PAM序列,很大程度局限了基因编辑靶点的选择性。扩展Cas的识别范围是当前对CRISPR/Cas9系统(尤其是碱基编辑系统)优化改良的重要方向。利用不同物种Cas9蛋白及其突变体等可在一定程度上扩宽CRISPR工具的打靶范围。例如,应用最广泛的化脓性链球菌Cas9 (SpCas9)识别DNA靶点下游保守的NGG PAM序列,金黄色葡萄球菌SaCas9识别NNGRRT PAM(Hua et al., 2018);SpCas9-NG、xCas9、SpG、SpRY(Ren et al., 2019; Xu et al., 2021)等SpCas9突变蛋白,经改造后具有不同或更为灵活的PAM兼容性,这在一定程度上拓宽了基因组编辑工具的靶向范围。然而部分Cas9蛋白及其突变体在扩宽了识别PAM的同时也存在一定程度编辑能力的降低。因此,开发识别PAM灵活和编辑效率高的Cas蛋白对于拓展CRISPR/Cas9系统的应用范围具有重要的意义。
近日,JIPB在线发表了中国农业科学院植物保护研究所周焕斌团队题为“CRISPR/Sc++-mediated genome editing in rice”(https://doi.org/10.1111/jipb.13166)的研究论文。该团队前期报道了来源于犬链球菌(Streptococcus canis)的ScCas9,在水稻中可通过识别NNG位点完成基因编辑,对NAG位点表现最好,但对NGG、NCG和NTG的编辑效率具有一定的位点依赖性,编辑效率低(Wang et al., 2020)。本研究通过对该ScCas9蛋白进行优化,利用获得的突变体Sc++对水稻基因组编辑技术进行优化升级。
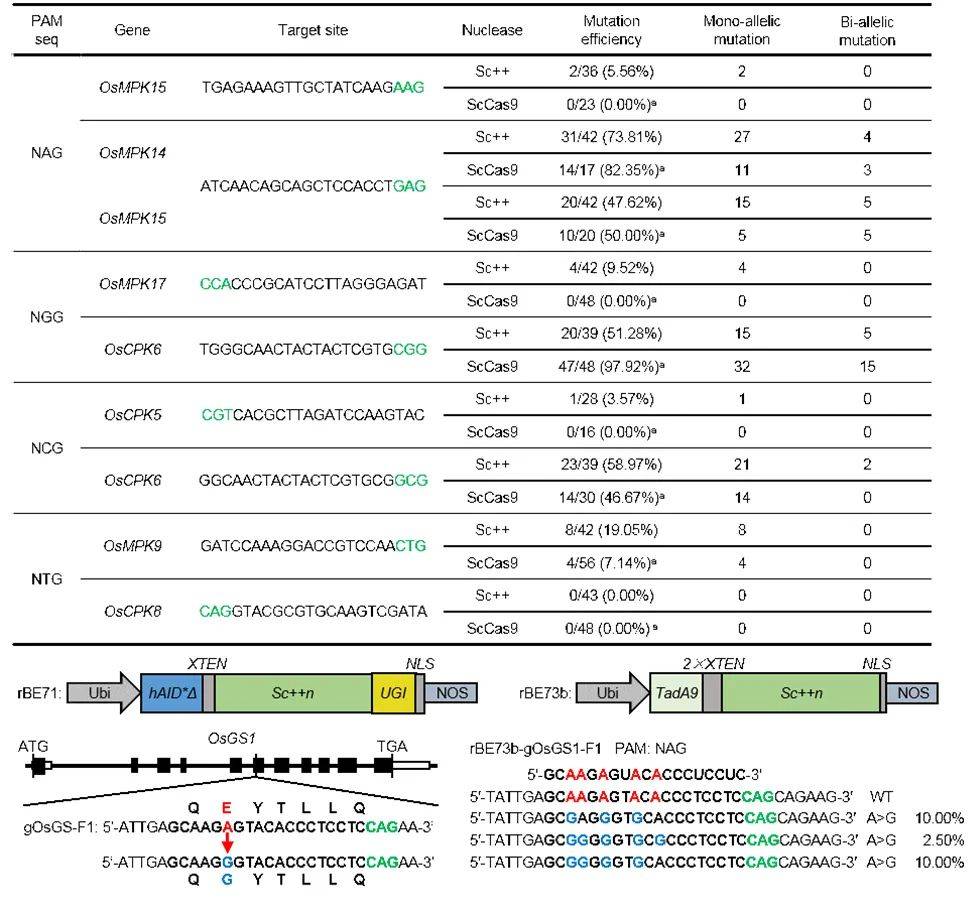
CRISPR/Sc++系统在水稻基因组编辑中的应用
研究发现,与ScCas9相比,Sc++核酸酶具有更广泛的基因组编辑功能,在NNG PAM位点具有较高的编辑效率。据此,该团队基于Sc++和高效的胞嘧啶脱氨酶hAID*Δ以及腺嘌呤脱氨酶TadA9开发了水稻胞嘧啶碱基编辑器rBE71和腺嘌呤碱基编辑器rBE73b。水稻转基因材料的检测结果显示,rBE71和rBE73b均能通过识别NGG、NAG、NCG等PAM完成碱基编辑,效率分别高达95.74%、52.63%和50.00%,还产生了除草剂抗性基因OsGS1的新等位基因,为今后筛选具有潜在除草剂抗性的水稻提供了新种质。综上所述,CRISPR/Sc++系统有效地扩展了水稻基因组编辑工具应用范围,为后续基因组编辑衍生工具开发、水稻功能基因组学研究和分子设计育种提供了有力的理论指导和技术支撑。
中国农业科学院植物保护研究所科研助理马桂根硕士和博士生旷永洁为论文共同第一作者,周焕斌研究员为通讯作者。该研究得到了中国国家自然科学基金和中央级公益性科研院所基本科研业务费专项的支持。
参考文献:
1. Hua, K., Tao, X., Zhu, J. (2019) Expanding the base editing scope in rice by using Cas9 variants. Plant Biotechnol. J. 17: 499-504.
2. Ren, B., Liu, L., Li, S., Kuang, Y., Wang, J., Zhang, D., Zhou, X., Lin, H., and Zhou, H. (2019) Cas9-NG greatly expands the targeting scope of the genome-editing toolkit by recognizing ng and other atypical pams in rice. Mol. Plant 12: 1015-1026.
3. Wang, M., Xu, Z., Gosavi, G., Ren, B., Cao, Y., Kuang, Y., Zhou, C., Spetz, C., Yan, F., Zhou, X., Zhou, H. (2020) Targeted base editing in rice with CRISPR/ScCas9 system. Plant Biotechnol. J. 18: 1645-1647.
4. Xu, Z., Kuang, Y., Ren, B., Yan, D., Yan, F., Spetz, C., Sun, W., Wang, G., Zhou, X., and Zhou, H. (2021) SpRY greatly expands the genome editing scope in rice with highly flexible PAM recognition. Genome Biol. 22: 6.
来源:JIPB
原文链接:http://mp.weixin.qq.com/s?__biz=MjM5ODc5NzY4OQ==&mid=2650217078&idx=1&sn=0a83ba2f1eb785469e14826891fd616f
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn



