来源:BioArt
责编 | 兮
T-box核酸开关(T-box riboswitches)是革兰氏阳性细菌内广泛存在的非编码核酸基因调控元件,负责监控并且维护细胞内大多数氨基酸的代谢通路【1-3】。T-box核酸开关直接结合特异的转运核糖核酸(tRNA),判别该tRNA末端是否携带氨基酸,并据此调节自身构象实现对下游氨基酸代谢基因的调控【4-6】。该类核酸开关的一个有趣特性是它独特的模块化设计(modularity)。T-box核酸开关一般包括三个结构域,分别发挥不同的功能:1)5’端的Stem I 区域负责辨识tRNA的整体结构和它特异的反密码子(anticodon)【4,7】;2)3’端的氨基酸判别器【5,6,8】;3)多数T-box 核酸开关还有一个中间的功能和结构尚不明确的 Stem II结构域。
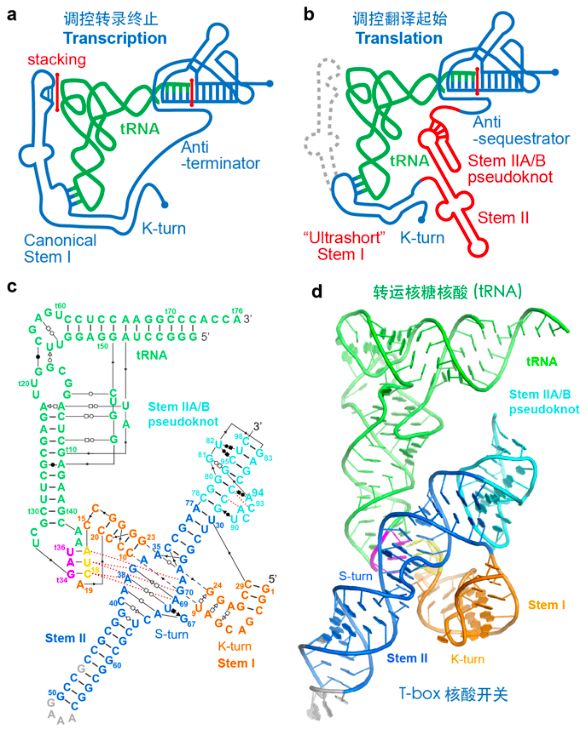
a.经典的T-box 核酸开关结合末端无氨基酸的tRNA配体,通过稳定抗终止子以调控转录终止【5】;b.新型的T-box核酸开关因缺失长链的Stem I区域无法结合tRNA的臂肘。Stem II区域(红色)补偿Stem I的缺失以恢复对tRNA的亲和力。此类T-box通过调整自身构象从而影响核糖体结合以实现对蛋白质合成的调控;c & d.诺卡氏菌(Nocardia farcinica)内调节异亮氨酸(Ile)的IleS T-box核酸开关和匹配tRNA的共晶体结构。
最早发现于1993年,超过一千个T-box核酸开关已经在多数革兰氏阳性菌包括大量病原菌内鉴定。早些发现的T-box核酸开关大多数在转录终止和抗终止水平上调控氨基酸相关基因。2015年,在放线菌门(Actinobacteria)包括结核病的病原菌结核分枝杆菌(Mycobacterium tuberculosis)内发现了多个新型T-box核酸开关【9,10】。这类新的T-box主要在蛋白质翻译起始水平上调控氨基酸代谢,并且相比传统调节转录的T-boxes有重要的架构上和机理上的区别【9,10】。
最近十年的T-box领域研究大多集中在一类经典的调控甘氨酸(glycine)的T-box核酸开关。此类T-box的特性是它们天然缺失结构复杂的Stem II 区域,并使用延长的的Stem I同时结合tRNA配体的反密码子区域和“臂肘”(elbow)区域。2015年以来新发现的调控翻译的T-box通常使用极短的Stem I区域,无法结合tRNA的臂肘。同时,这些T-boxes的功能依赖Stem II区域。最近有研究通过紫外线交联分析推测Stem II 区域直接辨识tRNA的臂肘附近区域从而增加对tRNA的亲和力以实现有效基因调控【9,10】。
2019年11月18日,美国国立卫生研究院(NIH)研究员张金伟研究组 (https://www-mslmb.niddk.nih.gov/zhang/zhanglab.html)在Nature Structure & Molecular Biology杂志上发表文章解析首个T-box核酸开关3’端的氨基酸判别器 (discriminator) 区域和tRNA的共晶体结构(2.66 Å分辨率),首次揭示了核糖核酸辨识tRNA氨酰化(aminoacylation)的分子机制(详见BioArt报道:NSMB | 张金伟/赵华组共同揭示T-box核酸开关的核心机理)。
2019年12月2日,张金伟研究组再次在Nature Structure & Molecular Biology杂志上发表长文文章High-affinity recognition of specific tRNAs by an mRNA anticodon-binding groove,使用X射线共晶体学和生物物理分析技术首次揭示了新型调节蛋白质合成的诺卡氏菌(Nocardia farcinica)T-box 核酸开关辨识tRNA和调控蛋白质合成的结构机制。

这类T-box核酸的Stem II区域并不像之前报道的结合tRNA的臂肘区域9,10【9,10】,而是通过与极短的Stem I对接共同构建出一个辨识并紧密结合tRNA 反密码子的新型功能元件。Stem II区域使用它中部保守的一个S-turn(S形回转)结构从侧面桥接以稳定Stem I密码子(codon)与tRNA反密码子(anticodon)之间的三个关键的碱基对。S-turn区域的多个单碱基点突变严重削弱了对tRNA的结合力,效果超过1000倍。Stem II不仅有效的增加了T-box结合tRNA配体的整体亲和力,这些的相互作用在很大程度上决定了对T-box密码子和特异tRNA的进化选择。
除了少数调控甘氨酸(glycine)的T-box核酸开关以外,大多数T-box核酸开关都具备这一Stem II 区域。因此对这一区域的结构和功能上的阐明延伸并深化了我们对T-box核酸开关的了解,并将有助于以T-box核酸开关为靶点的新一代抗生素药物的设计和研发。并且,此项研究提供了一个理解区域组织复杂,结合模式多元化,并依赖结构特异性的核糖核酸相互作用的范例。此研究工作主要由张金伟研究组博士后Krishna Suddala完成。

a.Stem I 区域的密码子(codon)与tRNA的反密码子(anticodon)形成3个碱基配对;b.Stem II 区域从侧面桥接从而稳定Stem I与tRNA的碱基配对;c.Stem II 使用S-turn (S形回转)结构在三个碱基配对的层面的分别加强极短Stem I与tRNA的相互作用,从而达到对tRNA足够的整体亲和力。
原文链接:
https://doi.org/10.1038/s41594-019-0335-6
参考文献
1.Grundy, F.J. & Henkin, T.M. tRNA as a positive regulator of transcription antitermination in B. subtilis. Cell 74, 475-482 (1993).
2.Zhang, J. & Ferre-D'Amare, A.R. Structure and mechanism of the T-box riboswitches. Wiley Interdiscip Rev RNA 6, 419-33 (2015).
3.Suddala, K.C. & Zhang, J. An evolving tale of two interacting RNAs-themes and variations of the T-box riboswitch mechanism. IUBMB Life 71, 1167-1180 (2019).
4.Zhang, J. & Ferré-D'Amaré, A.R. Co-crystal structure of a T-box riboswitch stem I domain in complex with its cognate tRNA. Nature 500, 363-366 (2013).
5.Li, S., Su, Z.,Lehmann, J., Stamatopoulou, V., Giarimoglou, N., Henderson, F. E., Fan, L., Pintilie, G. D., Zhang, K., Chen, M., Ludtke, S. J., Wang, Y. X., Stathopoulos, C., Chiu, W., & Zhang, J. Structural basis of amino acid surveillance by higher-order tRNA-mRNA interactions. Nat Struct Mol Biol (2019).
6.Battaglia, R.A., Grigg, J.C. & Ke, A. Structural basis for tRNA decoding and aminoacylation sensing by T-box riboregulators. Nat Struct Mol Biol (2019).
7.Grigg, J.C. & Ke, A. Structural Determinants for Geometry and Information Decoding of tRNA by T Box Leader RNA. Structure 21, 2025-32 (2013).
8.Zhang, J. & Ferré-D’Amaré, A.R. Direct evaluation of tRNA aminoacylation status by the T-box riboswitch using tRNA-mRNA stacking and steric readout. Mol Cell 55, 148-55 (2014).
9.Sherwood, A.V., Frandsen, J.K., Grundy, F.J. & Henkin, T.M. New tRNA contacts facilitate ligand binding in a Mycobacterium smegmatis T box riboswitch. Proc Natl Acad Sci U S A 115, 3894-3899 (2018).
10.Sherwood, A.V., Grundy, F.J. & Henkin, T.M. T box riboswitches in Actinobacteria: translational regulation via novel tRNA interactions. Proc Natl Acad Sci U S A 112, 1113-8 (2015).
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652478436&idx=6&sn=69cb9da3c9df74cd7518fbc0f1b4d9bf&chksm=84e20850b39581469a86781b88a4e38200a11975bba862a5acd496188dede646a11f19fd5d78&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

