来源:BioArt
CRISPR-Cas9是一种强大且广泛应用的工具,可用来产生精确的、可编程的基因编辑。基于CRSIPR的敲除或敲低可在多种细胞和生物体中进行系统性、全基因组遗传分析。Cas9蛋白结合gRNA,靶向gRNA互补的基因组位点,进而引入突变或沉默启动子。gRNA文库可用于产生一群细胞,而每个细胞表达一个独特的gRNA【1】。该单细胞的表型反映出每个gRNA所产生影响。影响细胞生存或增殖的gRNA所造成的表型较为直接,但生长是一个较为粗糙的表型,难以用于表征很多重要的生物学问题【2】。如果有新技术可以在不同群体中检测更为特异性和相关的表型,并将这些表型和单个的gRNA联系起来,那么将大大拓宽CRISPR的使用范围。一个重要基因的表达水平或一个重要蛋白的稳定性,可定义为分子表型,或可成为研究细胞生理学的一个敏感的测量工具。
2020年12月11日,来自加州大学伯克利分校的Nicholas T. Ingolia在Science杂志发表文章,CiBER-seq dissects genetic networks by quantitative CRISPRi profiling of expression phenotypes,将CRISPR干扰技术、报告基因标签化(barcoded)和测序结合研发出一种新技术CiBER-seq,可实现在细胞群体中应用整个gRNA的文库,同时在集中深度测序里将单个gRNA的表型区分开来。在酵母中使用CiBER-seq研究综合应激反应途径(ISR),发现可导致空载tRNA积累的遗传干扰能激活ISR报告基因的转录。tRNA的不足也可激活报告基因,但不依赖于空载tRNA感知器。在酵母中的成功应用,说明CiBER-seq可精确、全面地用于剖析遗传网络,是一种强大的工具。

CiBER-seq技术,就是将CRISPRi和报告基因的标签化进行结合。具体来讲,转染的质粒中,每一个标签条形码和一个gRNA、报告基因相对应,通过条形码将gRNA和报告基因的表达水平连接起来。当然条形码RNA的表达可能受到细胞数量或细胞活性的影响,选取一个管家基因启动子所驱动的条形码作为对照。
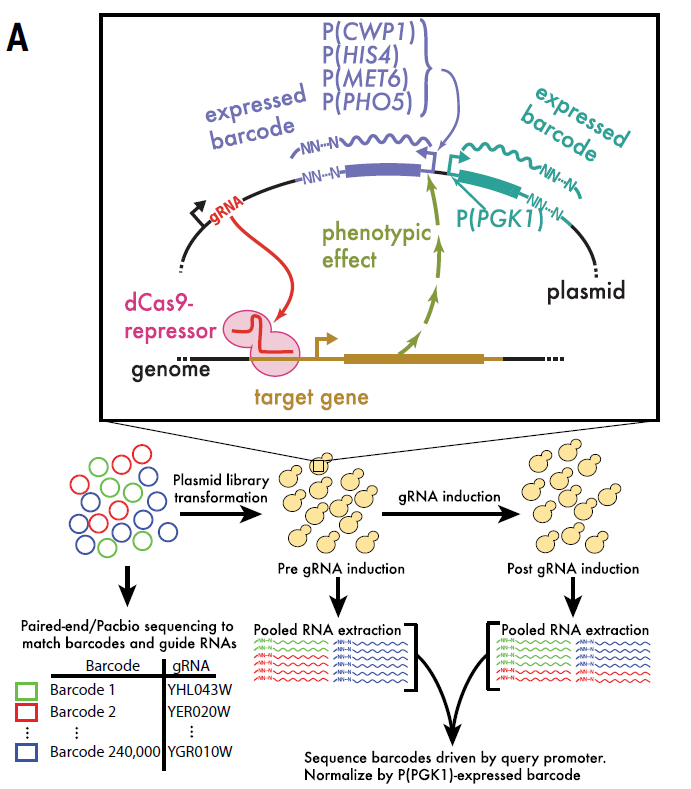
研究人员首先在酵母中应用CiBER-seq技术,鉴定出MET6、CWP1、PHO5、HIS4启动子(如P(HIS4))所驱动的报告基因的调控环路,这四种基因产物参与了一系列细胞内功能调节。四种启动子的调控环路都表现出与其已知调控和功能相符的表型特征,而且彼此之间独立且基本不重叠。例如,HIS4基因编码一种氨基酸生物合成酶,是酵母综合应激反应(ISR)的一个特征性转录靶点,这一高度保守的途径上调生物合成基因,以应对氨基酸饥饿期间出现的空载tRNA水平的升高。而CiBER-seq结果显示,敲低氨酰tRNA合成酶可激活(HIS4)启动子,空载tRNA可被Gcn2感知;但敲除靶向tRNA合成酶的gRNA则不能激活这一区域。敲低RNA聚合酶III导致tRNA消失,可同时激活(HIS4)和(MET6)启动子,而敲除Gcn2对其没有影响。为了进一步直接测定P(HIS4)的活性,将对照启动子P(PGK1)与P(HIS4)分别处于两个质粒中,然后进行单个的CiBER-seq。GO分析显示P(PGK1)和P(HIS4)的调控因子分属两个各自独立的特异的集合。单个的P(HIS4)CiBER-seq同样显示tRNA负载的缺陷和tRNA合成缺陷特异性影响这一启动子:有35个不同的gRNA靶向氨基酸合成通路可激活P(HIS4),其中有17/20个氨酰tRNA合成酶。
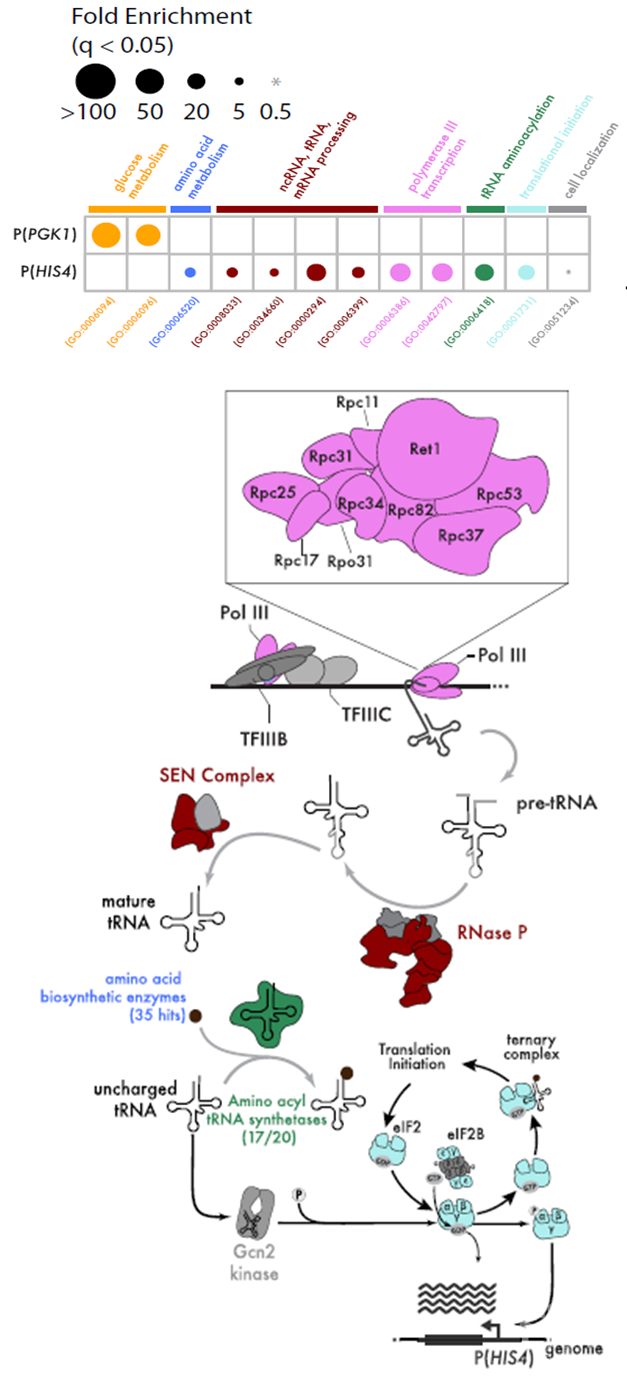
为了研究ISR过程中,典型的正向ISR信号转导元件如GCN2和GCN4的互作网络,研究人员利用3AT诱导P(HIS4)激活,在营养缺乏条件下可造成P(HIS4)启动子激活的gRNA、靶向RNA聚合酶II转录机器组分的gRNA均不能进一步增强P(HIS4)的激活,而敲低Gcn4降解因子PCL5可进一步增加3AT诱导的P(HIS4)激活。说明靶向氨基酸生物合成、tRNA负荷、转录和处理过程的gRNA都通过GCN4依赖性机制维持饱和的P(HIS4)转录。同时,tRNA不足所导致的P(HIS4)激活存在不依赖于GCN2的机制,tRNA招募缺乏导致的翻译延伸缺陷可直接诱发GCN4介导的转录反应。此外,敲除HTS1或RPC31能够诱导饱和的P(HIS4)转录,类似于3AT诱导。
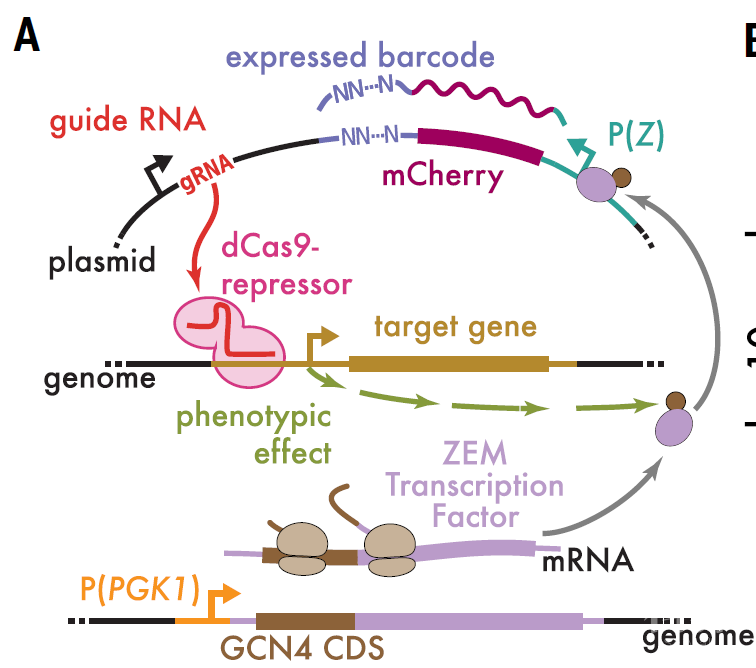
最后,研究人员在酵母中组成性表达GCN4和ZEM嵌合转录因子融合蛋白,从而区分出GCN4在蛋白水平和转录水平对P(HIS4)的分别调控:即gRNA靶向相对应的基因位点影响基因表达,基因表达如果对GCN4具有翻译后修饰调控,则影响条形码RNA的表达。实验显示,tRNA负荷或tRNA生物合成都对GCN4的翻译后修饰没有影响,而破坏SUMO化增加GCN4活性。突变GCN4的SUMO位点导致GCN4活性本底水平增加,但SUMO敲低不能进一步增加GCN4活性。利用GCN4 启动子和5‘引导序列驱动ZEM转录因子的表达,发现阻断tRNA生物合成和tRNA负荷的gRNA也可增加条形码表达,表明GCN4 5’引导序列是一种固有的翻译应激的生物感知器,且翻译调控是ISR激活的中心事件。

总的来说,研究开发了CiBER-seq工具,实现了在细胞群体中应用gRNA文库,利用集中测序就可以鉴定出单个gRNA的分子表型,不需要使用单细胞测序,简化了流程。同时,研究人员在酵母中应用此技术对综合应激反应途径进行研究,揭示出空载tRNA和tRNA缺乏对ISR途径的激活,证明CiBER-seq可提供一种强大的可广泛应用的研究遗传网络的工具。
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652514009&idx=4&sn=e59bdfe961bfbf57a39c905613686f6c
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn



