来源:宏基因组
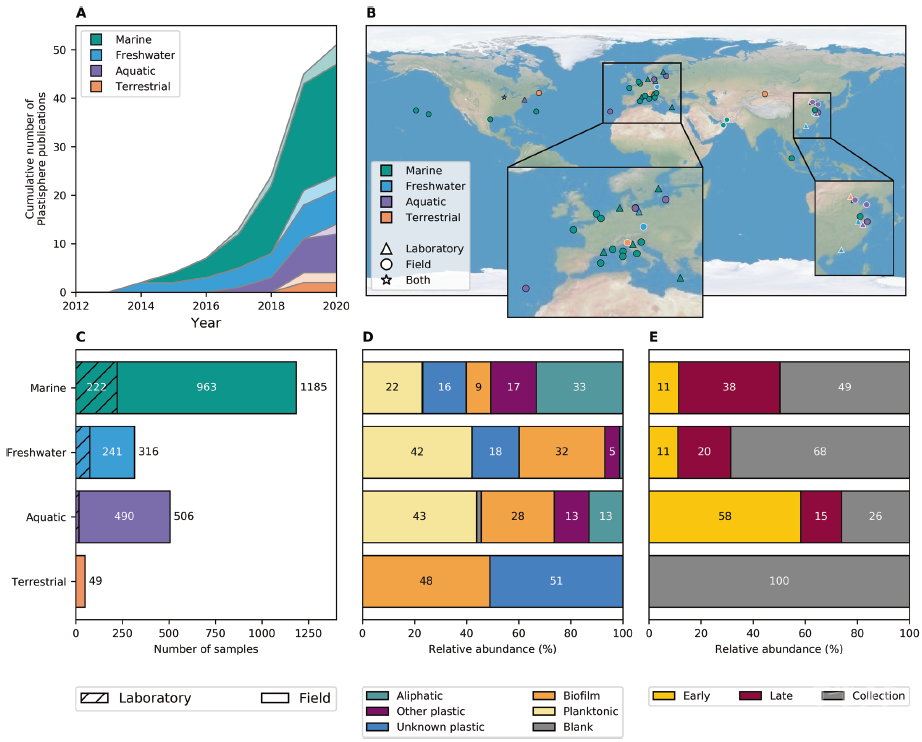
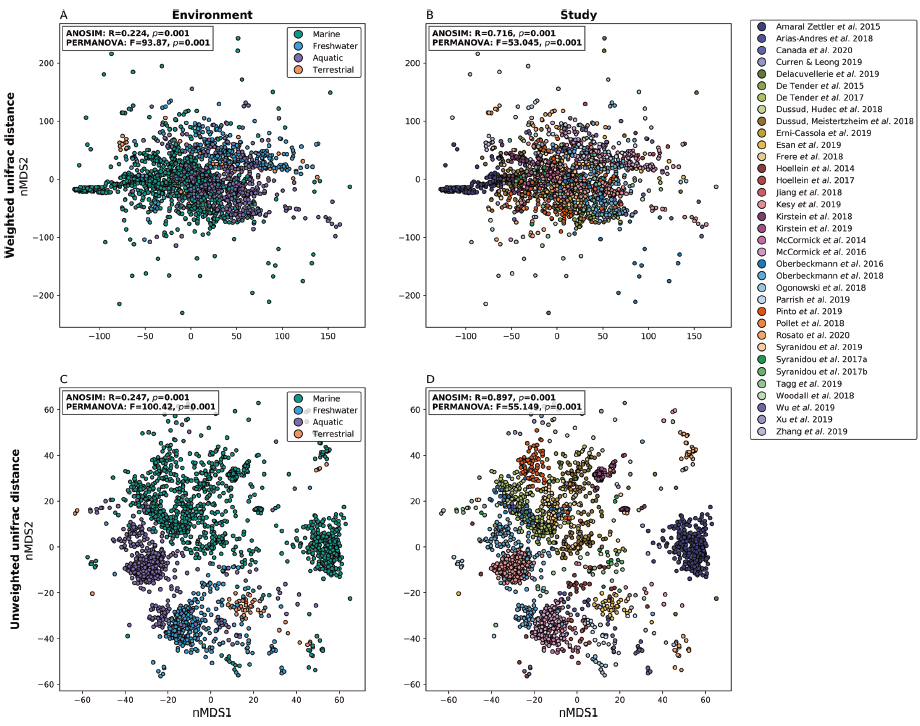
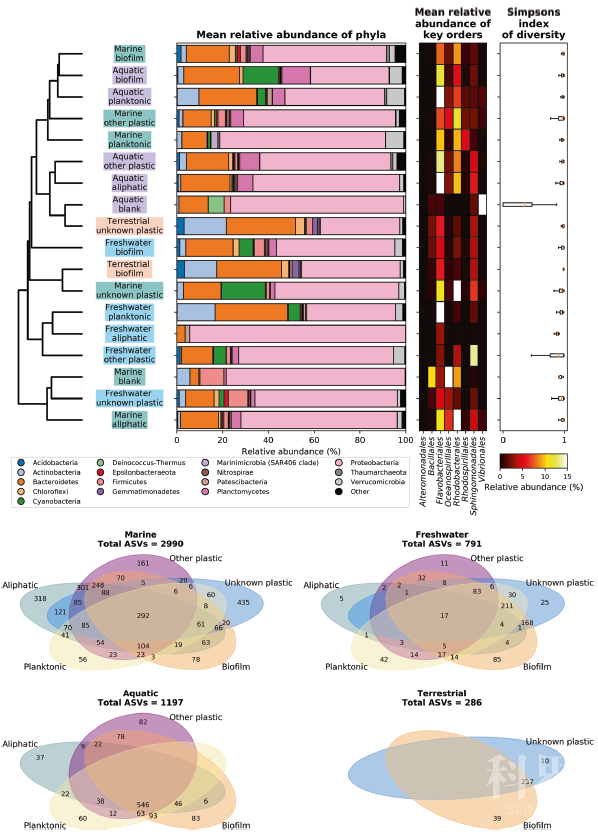
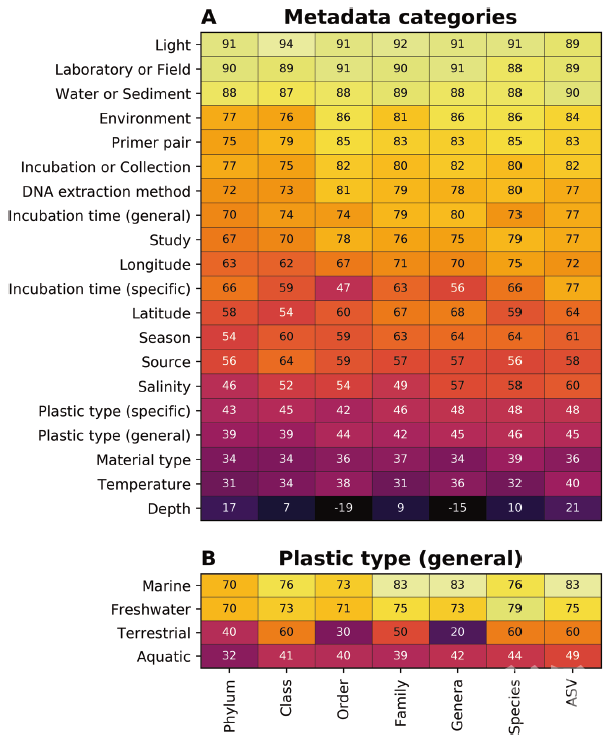
摘要塑料在全球生态系统中已经无处不在。定植在微塑料的生物膜(称为“塑料圈”)可能在塑料的迁移和生态效应中发挥着重要的作用。通过收集和重新分析35个研究2229个样品中所有的原始16S rRNA基因数据,作者们第一次对海洋、淡水、其他水生环境(例如:淡水养殖或者海水养殖)和陆生环境中的塑料圈进行meta分析。结果表明,训练后的随机森林模型可以区分不同的环境因素分组和不同实验设计。更为重要的是,与对照生物膜相比,不同塑料材质类型和群落演替的不同阶段都可以进行区分。Meta分析进一步证实,降解烃类的Oceanospirillales和Alteromonadales在塑料的含量始终比对照生物膜样品丰富,今后需要全面测试这些细菌对塑料的生物降解能力。最后作者对微塑料圈的研究提出了一些建议和思考。实验方法概述数据收集:作者于2020年1月5日在Web of Science核心合集和Science Direct中使用搜索词“Plastics plastisphere”,“Plastics microbial community”和 “Plastics microbial degradation”进行了文献检索。该搜索仅限于符合以下条件的研究:(i)在2010年至2019年之间发表;(ii)具有原始数据(iii)表征了在不可生物降解的塑料上形成的生物膜;(iv)使用Illumina NGS。作者将重点放在16S rRNA基因上,最终选择了35项研究的2,229个样本。如果没有盐度和温度信息,则根据一年中采样时这些区域的典型特征来估算。如果未指定任何光照信息,则假定为环境光。如果未确定塑料类型,则将其分类为“未知塑料”。数据整理:作者对原始数据进一步进行整理分类:(1)环境类型:分为陆地、海洋、淡水等;(2)室内还是野外研究;(3)是否有明确的孵育和收集时间:按照早期、晚期和未知时间进行整理;(4)来源于水还是沉积物/土壤;(5)微塑料类型:分为脂肪族塑料(PE或者PP),其他塑料(塑料含有官能团,比如:PET,PS和PVC),未知塑料;环境介质分为生物膜(对照基质,玻璃或者树叶),浮游细菌或者方法学空白。16S rRNA数据分析与处理:使用QIIME 2软件对每个16S rRNA原始数据进行分析,比对SILVA v132数据库进行细菌分类。删除reads少于2,000的样本,留下34个研究的2,056个样本,并使用SILVA v128数据库构建系统发育树。作者使用了几种归一化方法来解决不同样本测序深度上的巨大差异:(i)样本被抽平到2,000和转换为相对丰度;(ii)将样本转换为相对丰度;(iii)将样本转换为对数;(iv)将样本转换为中心对数比(CLR)。随机森林模型构建及不同环境共有物种的丰度:使用随机森林模型(离散或连续模型或分类模型)进行构建,分别有80%和20%的样本用于训练和测试。分别针对门、纲、目、科、属、种和ASV的分类学水平进行。以按环境分组的样本,计算处理之间共享的ASV。使用Wilcoxon Rank Sum测试确定在早期(≤7天)或晚期(> 7天)孵育时间之间的样品差异。结果简述 收集的样品信息:作者重新分析了从35项研究中获得的16S rRNA基因扩增子测序数据。迄今为止,所有的Plastisphere研究都集中于北半球,其中大部分研究是在欧洲及其周围进行的(图1)。除去所有reads小于2,000的数据后,剩余2,056个16S rRNA数据,其中海洋,淡水,水生和陆地环境中分别有1,185、316、506和49个数据。大部分样品都是未知孵育时间的野外采集样品(图1)。图 1 本文meta分析所选研究和样品概览归一化方法影响样品的生物学解释:在不同研究之间,每个样本的reads数量和分类学丰富度存在很大差异。因此,我们研究了几种不同的数据归一化方法对随机森林模型分类的准确性以及通过这些方法识别为重要的分类单元之间的一致性。使用数据抽平的方式构建的随机森林模型分类的准确性最低。基于转换为相对丰度的数据构建的模型最准确。数据抽平可以最有效地解决库大小的巨大差异,因此,作者选择降低随机森林分类的准确性,后续的分析中采用抽平数据。不同研究内部和研究之间的细菌多样性:Beta多样性分析(加权和未加权uniFrac距离)显示,按研究或环境分组的样本之间存在显着差异(PERMANOVA和ANOSIM p = 0.001;图2)。研究与环境之间的最大差异是由于生物体的丰度较低。一些基于实验室、从深海中收集样品或对没有包括V4 16S rRNA基因区域的研究与其他研究结果相似性较差。当将样本划分为环境和塑料类型,它们倾向于按环境分组,尽管并非在所有情况下都如此(图3)。除陆地环境,淡水浮游生物和水生生物膜对照组外,变形菌门占主导地位(相对丰度超过50%)。并且每种环境中的每种基质类型都具有独特的ASV(仅在该处理中相对丰度超过1%)。图2 样品之间基于有无权重uniFrac距离的nMDS图图3 样品分组中的微生物组成,多样性和共有的ASV环境变量对微生物群落影响最大而非塑料类型:作者为了分析环境变量和塑料材质的影响,针对环境变量(环境类型、地理位置、温度等)和塑料类型等不同因子,在门、纲、目、科、属、种、ASV等不同分类单元水平上构建了随机森林模型。使用ASV水平的微生物群落的模型最准确。光照条件(无论样品是在环境条件下还是在室内光照条件下孵育)是具有最高分类精度的变量指标(最高为94%;图4)。图4 随机森林模型的分类精度塑料圈中的潜在微塑料降解菌和病原菌:Oceanospirillales和Alteromonadales细菌在塑料样品中的丰度高于对照环境生物膜样品,而且Oceanospirillales在脂肪族塑料类型中丰度相对较高,而Alteromonadale在其他塑料样品类型中丰度较高。作者发现在塑料上丰度较高的一些病原菌类群:(1)鞭毛虫和比目鱼科生物的病原菌;(2)梭菌属人类病原菌、衣原体等。塑料圈微生物群落演替变化:淡水环境中的其他塑料和对照生物膜样品均显示出与海洋或淡水水体等不同的演替模式。海水环境中Oceanospirillales和 Alteromonadales细菌在脂肪族塑料制品生物膜形成早期丰度较高,而这种差异在晚期样品中消失。在淡水环境中,没有足够样本可用于配对的比较。
来源:meta-genome 宏基因组
原文链接:http://mp.weixin.qq.com/s?__biz=MzUzMjA4Njc1MA==&mid=2247493855&idx=3&sn=68ff53b7cf4967e0812906a7ee7f8a18
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
