来源:BioArt
细菌的抗生素耐药性是全球公共健康的一大威胁,替代抗生素类药物对抗细菌感染的方法亟待开发。噬菌体疗法是一种治疗细菌感染疾病的方法,通常不受细菌抗生素耐药性机制的影响【1】,因而在“超级细菌”横行的今天备受关注。近年来,不乏有抗生素治疗失败的病例能通过噬菌体疗法成功治愈【2,3】。传统的化学抗生素具有广谱杀菌活性,助长了细菌耐药性的产生,与之不同的是,噬菌体则对特定的菌株具有选择性。噬菌体的宿主选择依赖于与细菌表面受体的结合,进而识别宿主并引发感染【4】。
噬菌体所具有的受体识别特性,意味着细菌对噬菌体的抗性通过受体突变即可产生。因此,基于噬菌体生产的药物通常包含多种类的噬菌体,共同靶向同一宿主表面的多种受体,以降低单个噬菌体对受体的选择压力。然而,这些混合噬菌体药物制剂通常由生物学定义不清晰的噬菌体组成,因此很难追踪其药理学特性和免疫原性,这也限制了噬菌体作为抗菌药物的发展【5】。为了对抗细菌对于噬菌体药物耐受性的产生,合理地扩大噬菌体的宿主范围是目前亟待解决的问题。已有研究通过已知宿主域范围的噬菌体之间的杂交,或利用噬菌体的自然进化改变噬菌体的宿主范围,然而这些方法具有一定的局限性,或是缺乏对突变发生位点的控制,阻碍了我们对结构与功能对应关系的理解。
2019年10月3日,来自麻省理工学院(MIT)的Timothy K. Lu教授团队,在Cell杂志上发表了题为Engineering Phage Host-Range and Suppressing Bacterial Resistance through Phage Tail Fiber Mutagenesis的文章。该研究中,作者开发了一种特异性的、高通量的筛选方法,能够迅速扩大噬菌体的宿主范围,减缓细菌的耐药性产生,为新一代噬菌体抗菌药物的开发提供了新的思路。

为了克服噬菌体宿主域有限这一技术难题,研究者对噬菌体基因组中宿主识别相关区域进行了定向诱变,以增加这些关键位点的可筛选序列范围。以噬菌体T3为研究对象,通过与宿主菌BL21的感染对抗实验,研究者发现了噬菌体进化受限,不能抑制细菌耐药性的产生,并证实了T3尾丝中,由gp17远端编码的环状结构是宿主范围决定域(Host-range-determining regions, HRDRs)。而后,作者分别针对这些环状结构,包括内侧环CD、EF和GH以及外侧环BC、DE、FG和HI,用PCR扩增的方式替换每一个密码子,构建噬菌体文库(图1),其中HI和BC环的基因突变文库中包含了多样性较高的噬菌体突变体
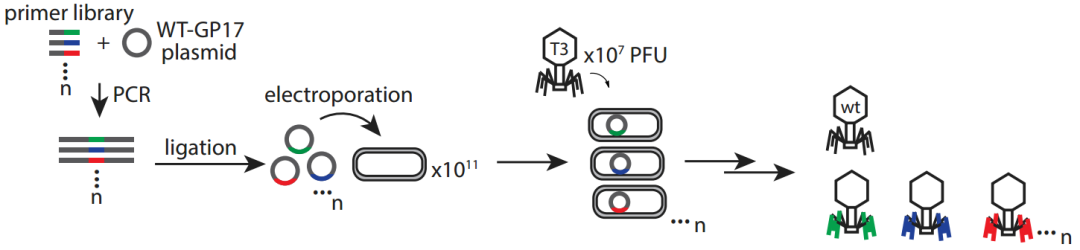
图1 噬菌体文库构建
在构建了针对不同环状结构突变的噬菌体文库后,作者通过感染筛选实验比较了这些文库与野生型WT T3在抑制宿主耐药性方面的表现,发现仅有HI环突变的文库阻止了感染后24h的细菌耐药性的产生。同时,上述结果也表明了这些文库中包含一些不发挥功能,或是不能识别宿主的部分。因此,作者进行三次重复平移实验,使得文库中能够感染抗性菌株的噬菌体大量产生,这也免去了进一步的文库富集过程。随后,作者从其中选择了10种确定的噬菌体组成混合制剂,与HI环突变文库相比,混合制剂虽然具有较低的噬菌体多样性,但其具有相似的感染能力,因此混合制剂颇具优势。
为了更好的理解工程改造后噬菌体(PB)抑制细菌耐药性产生的特性,作者还将其与自然产生的T3突变体(NM)在基因组、宿主范围以及杀菌效率方面进行了比较。研究最后,作者选择了具有广谱感染活性的PB10,以细菌感染皮肤后的小鼠为模型,证实了工程改造后的噬菌体在活体动物组织中依然具有宿主选择性和较高的活性。
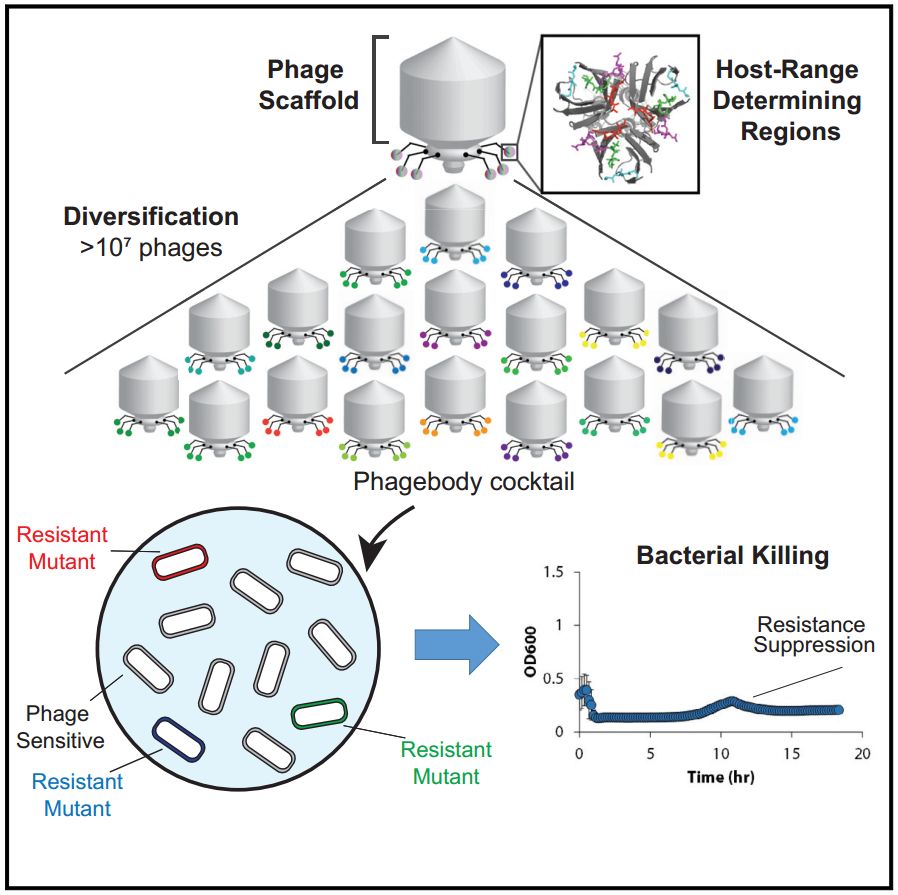
综上,该研究利用生物工程改造噬菌体,建立了宿主域更为广泛的噬菌体文库,并能够有效抑制噬菌体感染下的细菌耐药性产生。工程改造后的噬菌体具有已知的突变背景,且功能性噬菌体的规模较大,能够抵御细菌耐受性的产生。
值得注意的是,宿主细菌不仅能够通过表面受体突变产生噬菌体感染抵抗性,还能够通过其它机制,如顿挫感染(abortive infection)系统、CRISPR系统以及胞外多糖包裹以阻止受体结合的方式。因此,噬菌体疗法的未来研究方向将是阻碍细菌中噬菌体抗性的产生机制,以达到更高效、更安全的治疗效果。
原文链接:
https://doi.org/10.1016/j.cell.2019.09.015
参考文献
1. Mie dzybrodzki, R., et al. (2012). Clinical Aspects of Phage Therapy. Adv. Virus Res. 83, 73-121.
2. Dedrick, R.M., et al. (2019). Engineered bacteriophages for treatment of a patient with a disseminated drug-resistant Mycobacterium abscessus. Nat. Med. 25, 730–733.
3. Schooley, R.T., et al. (2017). Development and Use of Personalized Bacteriophage-Based Therapeutic Cocktails to Treat a Patient with a Disseminated Resistant Acinetobacter baumannii Infection. Antimicrob. Agents Chemother. 61, e00954-17.
4. Bertozzi Silva, J., et al. (2016). Host receptors for bacteriophage adsorption. FEMS Microbiol. Lett. 363, fnw002.
5. Cooper, C.J., et al. (2016). Adapting Drug Approval Pathways for Bacteriophage-Based Therapeutics. Front. Microbiol. 7, 1209.
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652477060&idx=4&sn=1cfff855cee6d4a5ef50c2ffe0c26664&chksm=84e20530b3958c26743226532c87eecd5358d67f35a8b6601541bd45ed4c4ff469af49aab0da&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn



