来源:BioArt
点评 | 左二伟(中国农科院农业基因组研究所)、周昌阳、杨辉 (中科院神经所)
撰文 | 木兰之枻
责编 | 兮
人类的致病遗传变异既有点突变又有碱基插入缺失突变【1】。2016年,哈佛大学David Liu实验室开发出新型单碱基编辑器CBE(Cytosine base editors),实现了C·G--T·A碱基对的自由转换【2】,2017年底他们又构建出ABE(Adenine Base Editor),可实现A·T--G·C碱基对的转换,这对众多点突变遗传病的治疗有重要意义,因而在基因治疗领域的应用前景相当广阔【3】(详见:突破丨Nature长文发表基因编辑最新成果——无需切割DNA也能自由替换ATGC)。不过,除了C·G--T·A和A·T--G·C碱基转换,对其它类型的碱基突变以及碱基的插入缺失突变,目前依然缺乏有效的研究工具:传统的同源重组修复(HDR)需要外源的双链/单链DNA模板,系统复杂且效率低下,这极大地限制了相关工作的开展,因此开发更加高效且广谱的精准基因编辑工具迫在眉睫。
2019年10月21日,哈佛大学David Liu实验室在Nature杂志上发表了题为Search-and-replace genome editing without double-strand breaks or donor DNA的论文。文章开发出了全新的精准基因编辑工具PE (Prime Editors),新工具PE无需额外的DNA模板便可有效实现所有12种单碱基的自由转换,而且还能有效实现多碱基的精准插入与删除(最多可插入44bp的碱基,可删除80bp的碱基),这一全能性的工具为基因编辑领域带来了重大变革。

新工具PE是以CRISPR-Cas9系统为基础,在两方面加以改造:首先是改造单链引导RNA (sgRNA),其3’末端增加了一段RNA序列,新获得的RNA被称作pegRNA;第二则是将Cas9切口酶(H840A突变型,只切断含PAM的靶点DNA链)与逆转录酶融合获得新的融合蛋白。pegRNA的3’端序列有双重角色,一段序列作为引物结合位点(PBS),与断裂的靶DNA链3’末端互补以起始逆转录过程,另一端序列则是逆转录的模板(RT模板),其上携带有目标点突变或插入缺失突变以实现精准的基因编辑(图1)。

图1 改造后的pegRNA结构
基因编辑工具PE的基本原理如图2所示,首先是在pegRNA的引导下,Cas9 H840切口酶切断含PAM的靶点DNA链,断裂的靶DNA链与pegRNA的3’末端PBS序列互补并结合,之后逆转录酶发挥功能,沿RT模板序列开始逆转录反应。反应结束后DNA链的切口处会形成处在动态平衡中的5’-和3’-flap结构,其中3’flap结构的DNA链携带有目标突变,而5’flap结构的DNA链则无任何突变。细胞内5’flap结构易被结构特异性内切酶识别并切除,之后经DNA连接和修复后靶位点处便实现了精准的基因编辑。
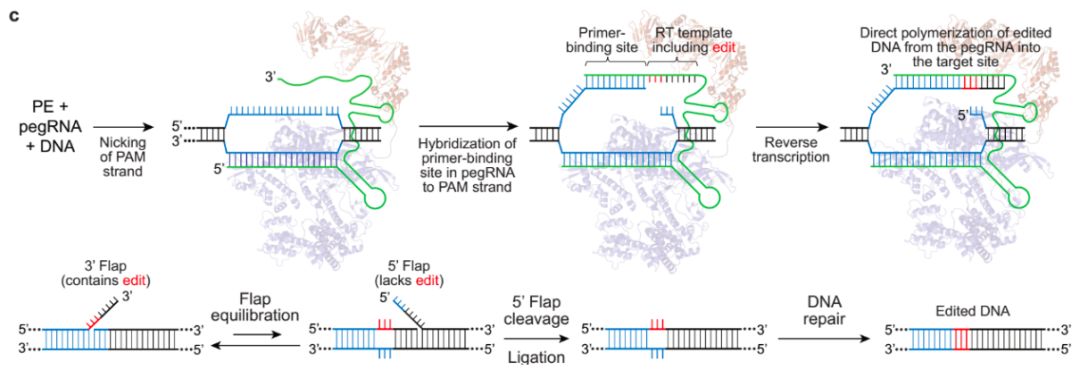
图2 基因编辑工具PE的基本原理
在经体外验证和酵母中的验证之后,研究者将野生型的鼠白血病病毒(M-MLV)逆转录酶融合在Cas9 H840切口酶的C末端,构建出了第一代精准基因编辑工具PE1,在293T细胞中PE1的点突变效率为0.7~5.5%, 碱基的增加/删除效率则为4~17%,依然有更大的提升空间。之后研究者通过优化M-MLV逆转录酶得到第二代编辑工具PE2,其点突变效率和碱基的增删效率较PE1有两倍以上的提高。
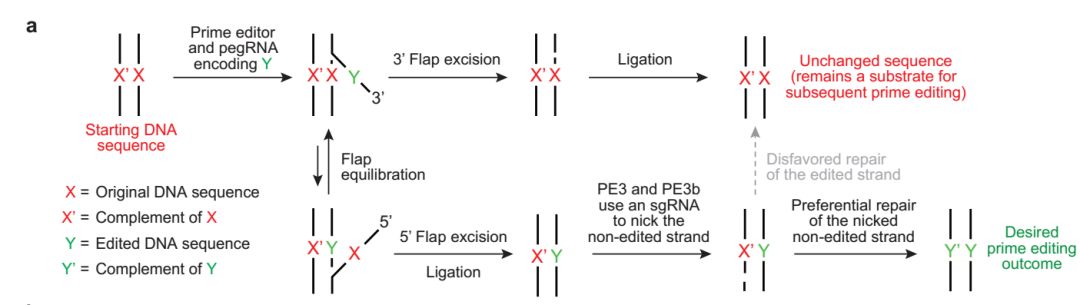
图3 PE3和PE3b的原理图:PE3增加的sgRNA识别位点是未编辑的基因组DNA,PE3b增加的sgRNA只识别编辑后的基因组DNA。
PE1/2系统只编辑双链DNA的一条链,另一条非编辑链需进一步的DNA修复以完成精准编辑。传统上,通过Cas9切口酶切断非编辑链可以有效提高该链的修复效率。为此研究者在PE2的基础上,增加可切断非编辑链的sgRNA,最终获得新的PE3和PE3b系统(图3)。新系统的编辑效率较PE2提升了近3倍,在293T细胞中的最高编辑效率可达78%。当然,由于使用了两条sgRNA,PE3的随机插入缺失(Indels)风险也随之提高,这是PE3未来需要加以改进的不足之处。
研究者对不同的工具进行比较后发现,与单碱基编辑工具CBE、ABE相比,PE3/3b的单碱基编辑在效率上略有不如,但能实现更精准的编辑;而与传统的HDR相比,PE3/3b有着更高的编辑效率和更低的Indels风险。此外,PE3/3b系统在U2OS、K562、HeLa三种细胞系以及小鼠皮层原代神经元中均能发挥精准编辑效果,这表明新系统有着广泛的适用性。
总体而言,本研究开发的新工具PE是精准基因编辑领域的重大突破,在单碱基随意转换和小片段多碱基的增删方面潜力巨大,这将极大的推动生物医学的基础研究和临床基因治疗研究。
专家点评
左二伟(中国农科院农业基因组研究所)、周昌阳、杨辉(中科院神经所)
大约有75000人类基因组位点和遗传疾病有关【4】。随着基因编辑快速发展,直接修改基因组治疗遗传疾病带来了可能性。使用CRISPR/Cas9基因编辑系统在致病位点产生DNA双链断裂,利用细胞的NHEJ或者HDR DNA修复通路,已经在动物模型上实现了治疗的人类遗传疾病的目的【5】。但是在大量细胞中同时诱导产生DNA双链断裂,有可能导致基因组的异位,倒位【6】,激活p53信号【7】等潜在问题,损伤基因组。为了消除这些不利因素,基因编辑大牛David liu与同事将失去催化活性的Cas9与脱氨酶融合,在sgRNA指导下,实现了对相应位点的碱基替换,建立了单碱基编辑系统【8,9】。根据融合的脱氨酶不同分为胞嘧啶单碱基编辑系统和腺嘌呤单碱基编辑系统,可以不产生DNA双链断裂的情况下将基因组中C转变成T, 或者将A转变成G。较高安全性和高效性促使单碱基编辑系统问世以来迅速在应用于动物植物以及人类基因组的编辑研究,并且利用动物模型研究治疗遗传疾病的探索。然而,最近多个实验室报道,单碱编辑工具会导致严重的基因组【10,11】和转录组【12,13】范围内的脱靶,虽有一些改良版本,可以消除转录组范围内的脱靶【12,14】,但是单碱基编辑系统编辑窗口比较窄,而且仅能编辑C-T, T-C, A-G, G-C,不能做C-A, C-G, G-C, G-T, A-C, A-T, T-A和T-G,也不能做插入替换等编辑,这些问题大大局限了单碱基编辑工具在遗传疾病治疗的应用范围。
近日,David liu 团队在Nature杂志发表了题为“Search-and-replace genome editing without double-strand breaks or donor DNA ”的研究论文,建立了一种被称为Prime editing的基因编辑新系统。在不产生DNA双链断裂,不使用DNA模版的情况下,可以在酵母和哺乳动物细胞靶位点高效产生DNA插入、删除和任意单碱基的替换。
他们将nCas9(H840A)与M-MLV逆转录酶形成融合蛋白,基因组结合序列、骨架序列、新的遗传信息以及单链DNA结合序列共同组建了pegRNA。在pegRNA指导下融合蛋白结合到基因组特定序列,nCas9使用单链切割活性,切开pegRNA非互补链。pegRNA携带的单链DNA结合序列与被切开的非互补链按照碱基匹配规律结合,单链DNA暴露一个3’-自由羟基,逆转录酶与之结合,按照碱基匹配规律以RNA为模版合成DNA。随后,合成后的DNA整合到基因组中,实现了靶向位点高效产生DNA的插入、删除和单碱基替换完成基因编辑的过程,构建了被称为PE1的系统。在PE1的基础上David liu团队又修改了M-MLV逆转录氨基酸序列(D200N+L603W+T330P+T306K+W313F),改善了热稳定性,逆转录连续性以及DNA:RNA结合紧密性,提高了编辑效率,建立了PE2系统。在PE2基础上,在pegRNA下游50bp附近,引入另一条与pegRNA方向相反的sgRNA,达到分别切割非互补链的目的,建立PE3系统,进一步提高了编辑效率。
Prime editing基因编辑系统与以往基因编辑系统相比避免了DNA双链断裂的产生、提高了基因编辑效率,拓展适用范围。然而Prime editing组成构件太大也限制了其在体内的临床应用。此外,逆转录酶作为主要构成要件,在细胞中过量表达,其安全性仍然是一个需要考虑的问题。
原文链接:
https://www.nature.com/articles/s41586-019-1711-4
制版人:珂
参考文献
1. Rees, H.A. & Liu, D.R. Base editing: precision chemistry on the genome and transcriptome of living cells. Nat Rev Genet (2018).
2. Komor AC, Kim YB, Packer MS, Zuris JA, Liu DR. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature 533, 420-424 (2016).
3. Gaudelli, N.M. et al. Programmable base editing of A*T to G*C in genomic DNA without DNA cleavage. Nature 551, 464-471 (2017).
4. Landrum, M. J. et al. ClinVar: public archive of interpretations of clinically relevant variants. Nucleic Acids Res 44, D862-D868, doi:10.1093/nar/gkv1222 (2016).
5. Jinek, M. et al. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science 337, 816-821, doi:10.1126/science.1225829 science.1225829 [pii] (2012).
6. Kosicki, M., Tomberg, K. & Bradley, A. Repair of double-strand breaks induced by CRISPR-Cas9 leads to large deletions and complex rearrangements. Nat Biotechnol 36, 765-771, doi:10.1038/nbt.4192nbt.4192 [pii] (2018).
7. Haapaniemi, E., Botla, S., Persson, J., Schmierer, B. & Taipale, J. CRISPR-Cas9 genome editing induces a p53-mediated DNA damage response. Nat Med 24, 927-930, doi:10.1038/s41591-018-0049-z10.1038/s41591-018-0049-z [pii] (2018).
8. Gaudelli, N. M. et al. Programmable base editing of A*T to G*C in genomic DNA without DNA cleavage. Nature 551, 464-471, doi:10.1038/nature24644 nature24644 [pii] (2017).
9. Komor, A. C., Kim, Y. B., Packer, M. S., Zuris, J. A. & Liu, D. R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage.Nature 533, 420-424, doi:10.1038/nature17946nature17946 [pii] (2016).
10. Zuo, E. et al. Cytosine base editor generates substantial off-target single-nucleotide variants in mouse embryos.Science 364, 289-292, doi:10.1126/science.aav9973 science.aav9973 [pii] (2019).
11. Jin, S. et al. Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice. Science 364, 292-+, doi:10.1126/science.aaw7166 (2019).
12. Zhou, C. et al. Off-target RNA mutation induced by DNA base editing and its elimination by mutagenesis. Nature 571, 275-278, doi:10.1038/s41586-019-1314-0 10.1038/s41586-019-1314-0 [pii] (2019).
13. Grunewald, J. et al. Transcriptome-wide off-target RNA editing induced by CRISPR-guided DNA base editors. Nature 569, 433-437, doi:10.1038/s41586-019-1161-z10.1038/s41586-019-1161-z [pii] (2019).
14. Grunewald, J. et al. CRISPR DNA base editors with reduced RNA off-target and self-editing activities.Nat Biotechnol 37, 1041-1048, doi:10.1038/s41587-019-0236-610.1038/s41587-019-0236-6 [pii] (2019).
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652476819&idx=1&sn=5639017bc8be6e13c9c3e5f004e4e53a&chksm=84e20627b3958f3189ee459ee2277dbfcb9199eed2c708b929f8fb8f76bd6168946fc14c9180&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn