来源:BioArt
基因编辑是指在活体基因组中进行DNA插入,删除或者突变的一项技术,它还有可能导致染色体重排,易位或者替换【1,2】。测序技术可以识别和检测这些变化,但是,却做不到实时动态监控。传统的荧光原位杂交技术(Fluorescent in situ hybridization,简称FISH)需要样本DNA发生变性,而荧光标记的cas9技术的样本则需要固定【3,4】。随着基因组研究的逐渐深入,学界亟需一种可以在活细胞中实时动态观测基因变化的技术。
2019年9月6日,来自斯坦福大学的Lei S. Qi团队在Science杂志上发表了题为 CRISPR-mediated live imaging of genome editing and transcription 的文章,报道了一种命名为“CRISPR活细胞荧光原位杂交技术(CRISPR LiveFISH)”的活细胞成像新技术。这种技术实现了在活细胞中实时观测单个细胞的基因组DNA和RNA片段,进而实时追踪基因组编辑、转录、重排和功能变化。

如下图所示,作者首先将等量的dCas9-EGFP融合蛋白和荧光标签Cy3标记的先导RNA(gRNA)合称为荧光-核酸-蛋白复合物(Fluorescent-ribonucleoproteins,简称fRNP),这个复合物特异针对3号染色体上一段重复序列。接着,作者将其转入人类骨髓瘤细胞系U2OS。流式细胞术检测发现,转入4小时后,95%的gRNA荧光信号发生淬灭,但是,在3号染色体上,却发现了稳定且高强度的荧光信号,这表明,染色体上已经形成了稳定的dCas9-gRNA复合体。基于上述实验,作者构建了CRISPR LiveFISH技术。并将这一技术加以推广应用。
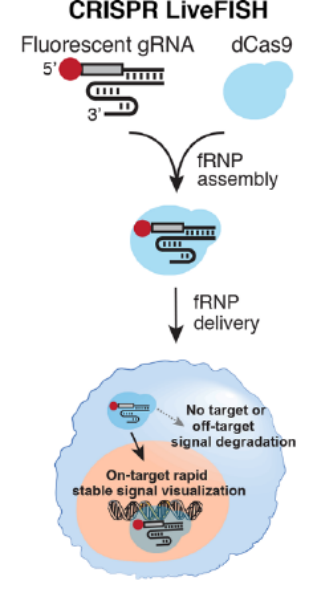
首先,应用CRISPR LiveFISH技术,可以在活细胞水平识别和定义染色体疾病。小儿帕套综合征(Patau syndrome),又称为13-三体综合征,患者具有3个13号染色体,从而导致严重的器官病变,体力智力低下,最终致命。作者设计fRNP特异针对13号染色体的重复序列,从而在患者单个活细胞中观察到3个重复的荧光信号,也就是3个13号染色体。
其次,应用CRISPR LiveFISH技术,可以实时观测DNA双链断裂(double-strand breaks,DSBs)。作者在U2OS细胞系中稳定表达带有DNA双链断裂感应子Apple的53BP1融合蛋白(即构建U2OS-53BP1-Apple 细胞系),然后共转入导致3号染色体PPP1R2位点双链断裂的基因编辑RNP(Cas9/gPPP1R2)和标记荧光信号的fRNPs。作者发现,基因组双链断裂处招募了大量53BP1蛋白,并且,这一高速且动态的过程持续了数小时之久。与此同时,作者还观察到诸多基因编辑细节。比如,53BP1蛋白的招募以及随后解离,表示此处双链断裂已然修复完毕。再比如同一位点53BP1反复招募解离,表示此处进行着活跃且反复的基因修复。还比如不同位点处于同一53BP1募集区(foci),则标志着发生同源重组。
再次,应用CRISPR LiveFISH技术,可以实时观测染色体易位。作者在上述细胞体系中同时转入两组基因编辑RNP,分别针对3号染色体的gPPP1R2位点和13号染色体的SPACA7位点。作者发现,这两个位点的基因活动极为相似,并且,这两组染色体从最初的保持一定距离,到缓慢的靠近,最终长时间相连,很可能是发生了染色体易位。随后,作者通过分子克隆技术,证实了此处的确发生了染色体易位。
最后,应用CRISPR LiveFISH技术,可以同时实时观测RNA和DNA。作者采用U2OS 2-6-3细胞系,此细胞系中含有一段LacO重复序列,并且,其下游连有多西环素(Doxycycline,简称Dox)诱导的MS2序列读码框架。作者同时转入针对LacO DNA的Cas9 gRNA和针对MS2 RNA的Cas13 gRNA【5,6】,发现,在Dox诱导下,DNA和RNA都被成功标记,并且,MS2逐渐聚集在LacO区域。
这篇文章是方法学研究的精品。作者综合了活细胞显微术,荧光原位杂交术和CRISPR基因编辑技术,创建了CRISPR活细胞荧光原位杂交技术(CRISPR LiveFISH)。这一技术可以实时检测单细胞中的DNA双链断裂,染色体易位,DNA-RNA相互作用等活细胞基因变化,并且,可以识别和定义染色体疾病。这一技术的亮点有两处,一是可以直接应用于活细胞,从而发现基因组活动的动态细节。二是通过与其它分子生物学手段的联合应用,相辅相成,可以更加高效深入的研究基因组的活动变化。
原文链接:
https://science.sciencemag.org/content/early/2019/09/04/science.aax7852
参考文献
1. P. S. Choi, M. Meyerson, Targeted genomic rearrangements using CRISPR/Cas technology. Nat. Commun. 5, 3728 (2014).
2. M. Kosicki, K. Tomberg, A. Bradley, Repair of double-strand breaks induced by CRISPR-Cas9 leads to large deletions and complex rearrangements. Nat. Biotechnol. 36, 765–771 (2018).
3. P. R. Langer-Safer, M. Levine, D. C. Ward, Immunological method for mapping genes on Drosophila polytene chromosomes. Proc. Natl. Acad. Sci. U.S.A. 79, 4381–4385 (1982).
4. W. Deng, X. Shi, R. Tjian, T. Lionnet, R. H. Singer, CASFISH: CRISPR/Cas9-mediated in situ labeling of genomic loci in fixed cells. Proc. Natl. Acad. Sci. U.S.A.112, 11870–11875 (2015).
5. S. Konermann, P. Lotfy, N. J. Brideau, J. Oki, M. N. Shokhirev, P. D. Hsu, Transcriptome Engineering with RNA-Targeting Type VI-D CRISPR Effectors. Cell 173, 665–676.e14 (2018).
6. 24. S. M. Janicki, T. Tsukamoto, S. E. Salghetti, W. P. Tansey, R. Sachidanandam, K. V. Prasanth, T. Ried, Y. Shav-Tal, E. Bertrand, R. H. Singer, D. L. Spector, From silencing to gene expression: Real-time analysis in single cells. Cell 116, 683–698 (2004).
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652474951&idx=6&sn=7ab5cb1987ca183c91318de388c14493&chksm=84e21df3b39594e50dfe4d578f007c1d7201eb029a8d53c027d6ef12f92de5f5dd7d08f88a0e&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn



