来源:BioArt
多细胞生物仅仅由单个细胞发育而来的全过程非常令人惊讶。在线虫中可以使用经典的谱系追踪实验来研究单细胞到多细胞的发生发育过程,并从中发现细胞谱系之间的差异以及功能表型方面的不同,但是这些研究结果也是取决于线虫本身的发育过程已经研究地非常透彻。更复杂的生物体的是如何精妙地调控的身体发育过程的还很不清楚。
为了进一步揭开哺乳动物早期胚胎发育过程,加州大学旧金山分校Jonathan S. Weissman研究组与MIT&Harvard Board研究所的Alexander Meissner研究组在Nature上合作发文Molecular recording of mammalian embryogenesis,为小鼠原肠胚发育过程的研究进一步提供了可用的工具。

单细胞RNA测序技术应用的如火如荼,对于揭开细胞种类的异质性、研究多种动物中的发育过程提供了重要的工具平台【1,2】(关于哺乳动物胚胎发育过程,BioArt在3月10日曾报道了两篇背靠背Nature文章,详见:背靠背Nature长文丨曹俊越博士等利用单细胞测序揭示胚胎发育路径)。最近,以CRISPR-Cas9为基础的技术也被用于记录细胞谱系发生【3】。但是这些研究编码多样性的产生很少,很大程度上限制了在其他组织和生命体中的应用。
那么理想的多细胞生物中发育过程的分子记录器需要具备什么样的特点呢?1)首先对于细胞表型方面的影响要降到最低;2)对于数以万计的细胞能够高通量记录信息;3)能够同时分析单细胞的功能信息;4)可用于记录大尺度时间线上的变化;5)在整个实验过程中连续产生多样性用于研究。因此,能够满足以上要点的技术需求随着人们想要进一步揭开生命发育过程中奥秘而与日俱增。
Weissman与Meissner研究组的工作希望建立一种方法能够同时记录细胞谱系的历史以及细胞的状态。为了达到该目的,他们使用Cas9产生双链断裂修复后造成可遗传的插入或者缺失突变。他们记录的是包含3个切割位点以及8碱基对的“整合条码”(Integration Barcode)205个碱基对的合成DNA。该合成DNA序列被嵌入组成型转录的荧光蛋白的3‘非翻译区,从而能够通过荧光从转录组中分析该实验结果。另外一个cassette编码了三个guide RNA,用来记录多种直接信号(图1)。此系统能够产生大量的可遗传修复结果和目标位点,并且可以通过整合条码对这些序列进行区分。这些guide RNA是他们通过筛选得到的,可以使用GFP报告基因在超过20天的时间对guide RNA靶标的活性进行监控,从而筛选具有广泛动态变化范围的系列。
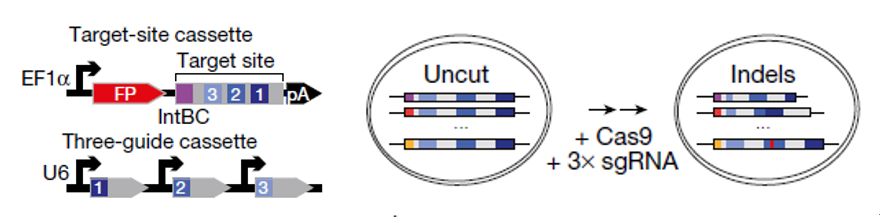
图1 “分子记录器”优化实验原理。
然后他们将优化后的技术运用在小鼠胚胎早期发育过程从而研究其中细胞命运的变化。将多个靶点整合到基因组中之后,他们又将组成型的Cas9-GFP编码的精子释放到卵母细胞之中开始启动切割。为了证明该技术的谱系追踪能力,他们对于胎盘、卵黄囊以及E8.5或者是 E9.5的小鼠胚胎中的靶点进行扩增,并且通过插入缺失标记的相似性来研究这三者之间的关系,从而评估该技术的实用性。通过该Cas9-GFP为基础的谱系追踪技术,作者们对于7个胚胎中的单细胞数据进行分析后发现,与细胞中的插入缺失突变标记相似。这些缺失突变标记并不会因为转入体内而受到影响,因此进一步确认了优化后的技术可以用于体内追踪细胞谱系的变化。
与此同时,作者们使用单细胞RNA测序技术对野生型小鼠的原肠胚形成过程中的细胞状态进行研究,发现引入的剪切并不会影响小鼠的正常发育过程。随后作者们利用单细胞测序结果系统发育重建策略对细胞谱系进行分类,从而确定他们建立的细胞谱系“追踪器”是否有效。通过单细胞测序的结果建立系统发育树后发现,细胞谱系发育距离越近,转录谱的相似度越高,该结果与细胞在发育过程中逐渐分化成特化的细胞类型相一致。同时也可以利用单细胞RNA测序的数据来建立一个全面的细胞命运图谱。
该研究中使用大量信息和持续记录的技术来展现了哺乳动物原肠胚形成过程细胞命运谱系示踪。通过将CRISPR-Cas9技术与单细胞RNA测序技术联用并进一步优化,揭开了哺乳动物发育过程中的“黑匣子”难题,为进一步回答更高阶的细胞组织的时空调控等问题打破了技术瓶颈。希望在未来能够在大尺度发育过程中的细胞命运决定的分子调控方面提供可用的工具和可供参考的思路。
参考文献
1. Han, X. et al. Mapping the Mouse Cell Atlas byMicrowell-Seq. Cell 173, 1307,doi:10.1016/j.cell.2018.05.012 (2018).
2. Wagner,D. E. et al. Single-cell mapping ofgene expression landscapes and lineage in the zebrafish embryo. Science 360, 981-987, doi:10.1126/science.aar4362 (2018).
3. Spanjaard,B. et al. Simultaneous lineagetracing and cell-type identification using CRISPR-Cas9-induced genetic scars. Nature biotechnology 36, 469-473, doi:10.1038/nbt.4124(2018).
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652469883&idx=1&sn=e2d5a52ef96d0c74ea6c4f44bc390e11&chksm=84e2e9cfb39560d9e62ca8ac3b8460a1abbe6ca50d0413bb22b008680ef0c6631270cbd5427b&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn



