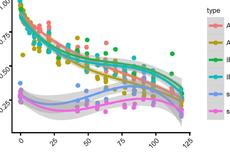
来源:宏基因组
我们将花时间把此文的原始代码整理并精讲,祝有需要的小伙伴能有收获。
本系列按原文4幅组图,共分为4节。本文是第4节,随机森林回归。
之前我们用了三篇文章,对随机森林的应用、分类、回归进行讲解和实战如下:
一文读懂随机森林在微生态中的应用
随机森林randomForest 分类Classification
随机森林randomForest 回归Regression
今天以图3中的一个子图,来实践一下冲击图的绘制。
前情提要
水稻微生物组时间序列分析
1模式图与PCoA
2a-相关分析
2b-散点图拟合
3-冲击图
先回顾一下图4的内容。
哪些菌可以作为生育时间的biomarkers?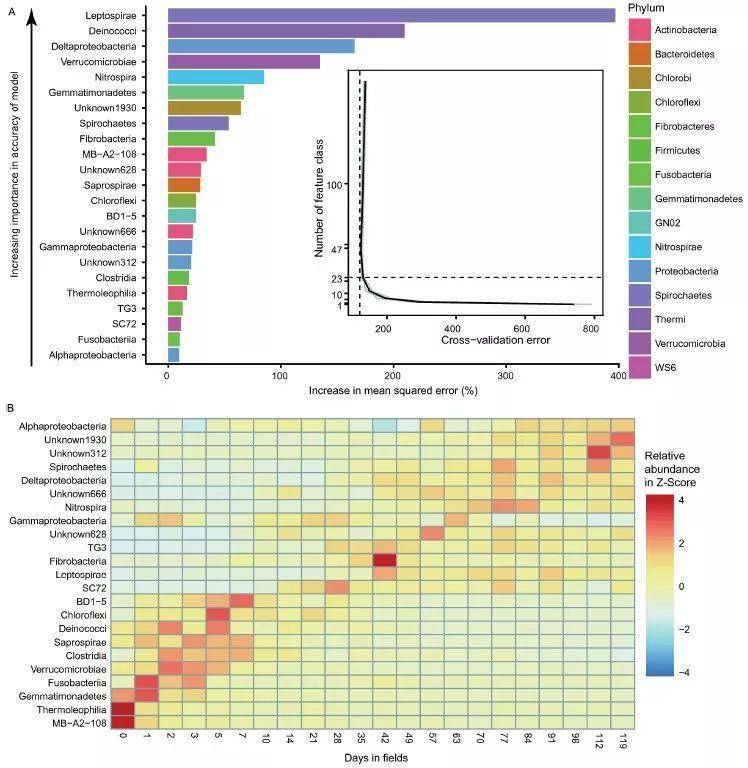
图4. 水稻生育期相关的微生物标记物(biomarkers)。
A. 采用随机森林方法在两地点的两品种样本中鉴定了23个纲与生育时间相关。其中按贡献度由大到小排序。其中的子图为交叉验证评估的结果。
B. 热图展示23个年龄相关的biomarkers相对丰度。
方法简介:本图A采用R语言的RandomForest包进行分析,结果采用ggplot2的柱状图进行可视化,biomarkers按贡献度由大到小排序,并进行交叉验证模型的准确度和biomarkers数量的选择依据。图B采用pheatmap展示每个时间点biomarkers的相对丰度均值,其中biomarkers按出现最高丰度的时间排序。
回归分析统计分析,主要基于两个表:OTU表和实验设计表,对于想进一步讨论分类级,别需要OTUs的物种注释文件。
这样基于这3个文件,可以制作出千变万化的统计分析图片,来作为论据支持你的文章(Story)。
时间序列做回归,主要是想建模来预测其它样品的生育时间。主要分为两部分,训练集建模,测试集验证。
我们主要有两个品种,种植在两个地点。这里先以A50建模,IR24验证的方案来演示。本实验较复杂 ,具体的方法会有多种组合。
绘制验证结果
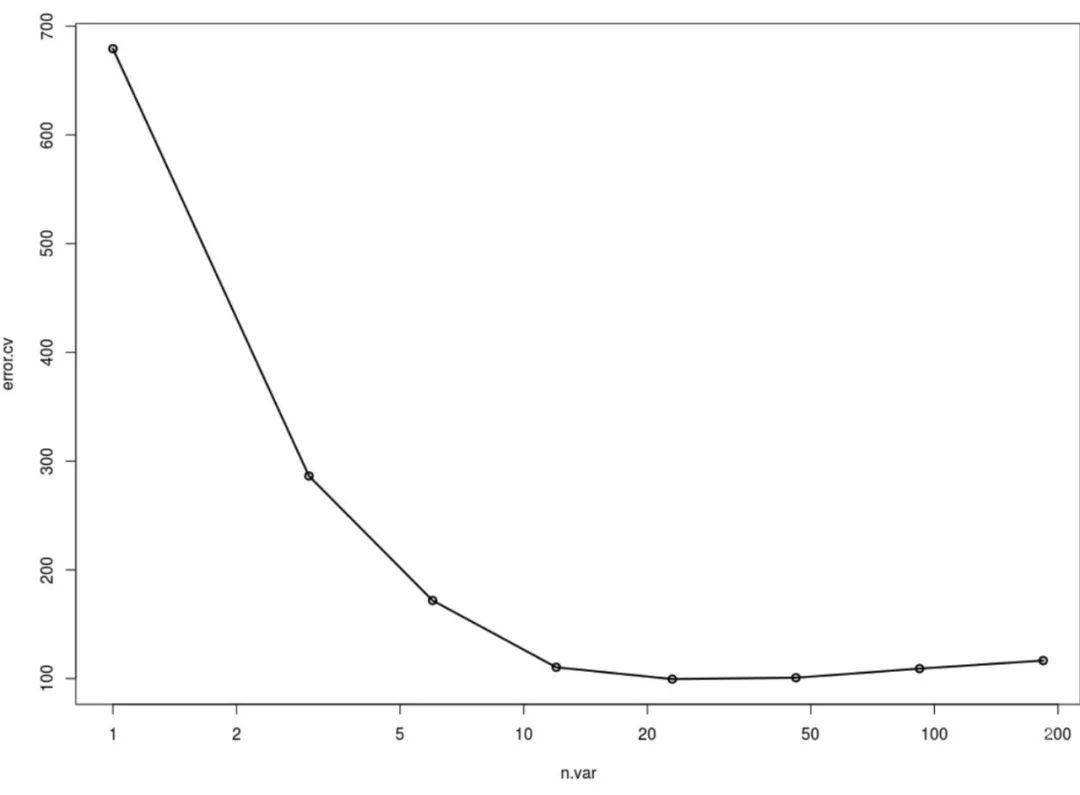

想绘制图中样式的图,可以使用imp的值,和名称中对应的物种注释进行数据筛选即可。这属于美化工作,下面开始,个人风格,仅供参考。
美化feature贡献度柱状图软件内部的varImpPlot可以快速可视化贡献度,简单全面,但发表还是要美美哒,美是需要代码的,就是花时间
基本思路同绘制Top 23 feature柱状图,按门着色,简化纲水平名字
来源:meta-genome 宏基因组
原文链接:https://mp.weixin.qq.com/s?__biz=MzUzMjA4Njc1MA==&mid=2247491271&idx=5&sn=f27daca017890271e2acab3ab8ebc05c&chksm=fab9f476cdce7d60fbf7a616f81ddd625c73dedc28aac77b4e017f4511eed1ff88d4b63edf71#rd
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn