来源:BioArt
原标题:Nat Biotech:CBE单碱基编辑器的Cas9非依赖型DNA脱靶风险评估及优化
撰文 | 木兰之枻
目前已知的人类致病遗传变异中,约58%都属于点突变,而约15%的致病点突变都属于T·A--C·G变异【1】。2016年哈佛大学David Liu实验室开发出新型单碱基编辑器CBE(Cytosine Base Editor),可实现C·G--T·A碱基对的转换,这对上述的点突变遗传病的治疗有重要意义,因而在基因治疗领域的应用前景相当广阔【2】。不过,2019年有研究通过全基因组测序分析指出,除了传统的Cas9非特异性结合导致的DNA脱靶风险之外,CBE系统还存在Cas9非依赖型的DNA脱靶风险【3-4】(详见:专家热评Science丨杨辉组/高彩霞组“背靠背”首次发现单碱基编辑系统存在严重脱靶效应),这极大地限制了CBE系统的应用。目前,CBE系统的Cas9非依赖型脱靶风险评估主要依赖于全基因组测序分析,该方法虽然全面,但耗时费力,花费不菲,难以用于规模化的评估和筛选,因而极大的限制了相关研究的开展,对CBE系统的优化和未来应用极为不利。
2020年2月10日,来自Broad研究所、哈佛大学化学系和化学生物系的David Liu实验室在Nature Biotechnology杂志上发表了题为Evaluation and minimization of Cas9-independent off-target DNA editing by cytosine base editors的论文。文章对多种CBE系统的Cas9非依赖型DNA脱靶风险进行了评估并加以优化。文章开发出较全基因组测序更为快速、便捷且经济的实验策略,并以此为基础对CBE系统的Cas9非依赖型DNA脱靶风险进行了有效评估;之后研究者还通过蛋白质工程等策略对CBE系统进行优化,有效地减少了Cas9非依赖型DNA脱靶风险的发生。
首先,研究者尝试通过细菌水平的利福平耐药实验来检测CBE系统的Cas9非依赖型DNA脱靶风险。其基本原理是,如若CBE系统中的胞苷脱氨基酶可诱发大肠杆菌中rpoB基因上的C-T突变,会导致大肠杆菌耐受利福平,之后根据耐药克隆的比例便可评估其Cas9非依赖型DNA脱靶风险。此外,研究者会同时在大肠杆菌中转入靶位点上携有T-C失活突变的氯霉素抗性基因表达载体,根据氯霉素抗性克隆的比例,便可同时评估CBE系统的碱基编辑活性。在证实了系统的有效性之后,研究者首先对CBE系统的三种组分胞苷脱氨基酶、dCas9及UGI与Cas9非依赖型DNA脱靶风险的关系进行分析,发现胞苷脱氨基酶和UGI是关键。这两者之中,胞苷脱氨基酶诱导利福平耐药的能力是UGI的上百倍,因而成为后续研究的重点所在。之后研究者发现APOBEC3G,AID,eA3A和APOBEC1的多种突变体如YE1等为基础的CBE系统能降低Cas9非依赖型DNA脱靶风险。此外,考虑到胞苷脱氨基酶的活性多存在序列偏好性,研究者还尝试用细菌胸苷激酶基因突变实验进行评估以保证结果的准确性并获得相似的结果。
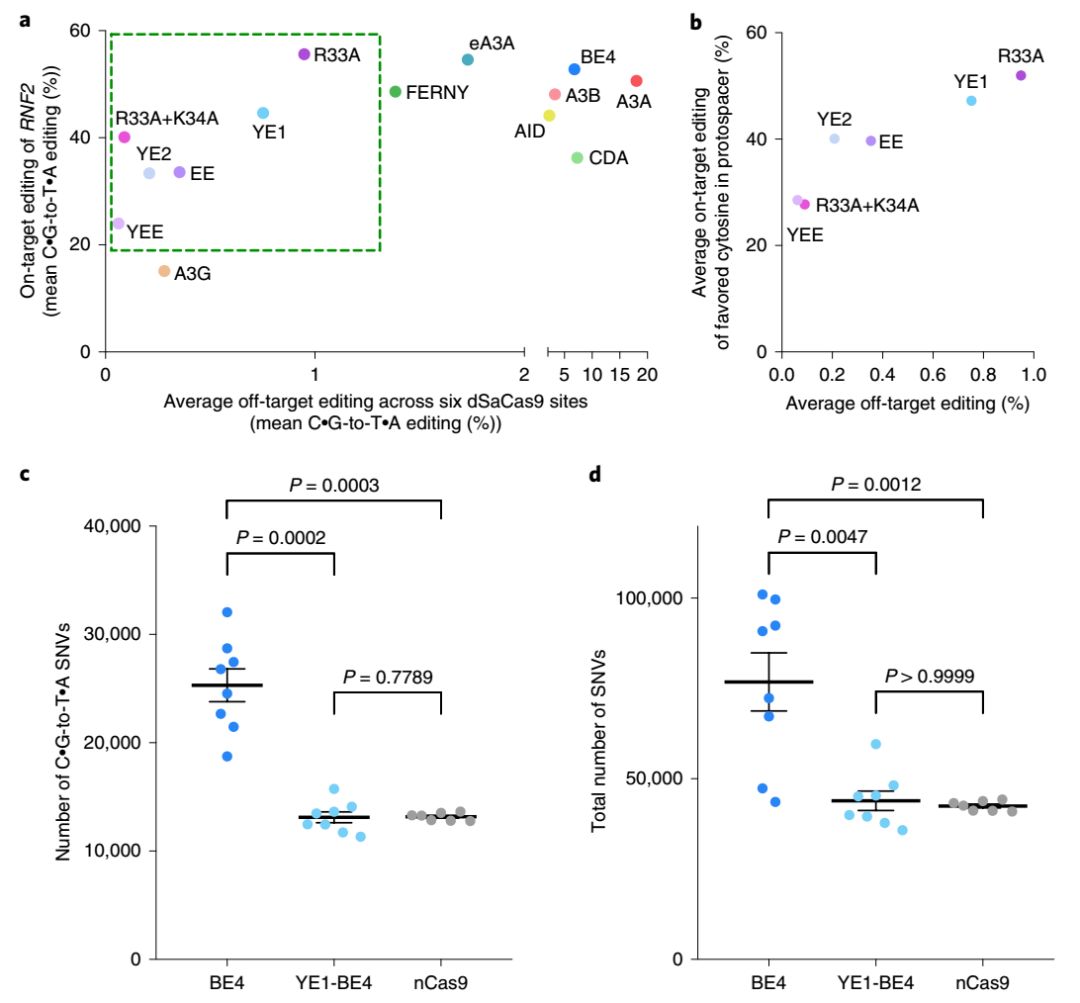
先前的研究和上述的细菌实验均证实,CBE系统的Cas9非依赖型DNA脱靶频率低于~0.1%,传统的定点扩增难以有效检测。为替代全基因组测序,研究者设计出可在人源细胞系中开展的R-loop实验方案,此方案可放大CBE系统在特定位点的Cas9非依赖型脱靶频率,之后便可通过定点扩增加以检测和评估。其基本原理是,通过不同类型的CRISPR系统在基因组特定位点诱导出单链DNA区域(ssDNA域),以“放大”该区域CBE系统的Cas9非依赖型胞嘧啶脱氨基频率,之后便可通过定向扩增和高通量测序加以检测。实际操作中,研究者在293T细胞中共转染SpCas9为基础的CBE系统及spsgRNA,失活型SaCas9(dSaCas9)及sasgRNA,之后检测sasgRNA靶位点的胞嘧啶脱氨基频率。通过该实验,研究者发现,APOBEC1的多种突变体如YE1、YE2、EE、YEE、R33A和R33A/R34A为基础的CBE系统有着最低的Cas9非依赖型DNA脱靶风险(图1a-b)。
根据以上结果,研究者推测,Cas9非依赖型DNA脱靶风险主要取决于胞苷脱氨基酶的催化效率,理想的CBE系统应当有着较低的催化效率,以保证在底物低浓度时处于失活状态(脱靶风险低)而在底物高浓度时方发挥催化功能(保证靶位点的编辑效率)。体外动力学研究证实,与野生型rAPOBEC1相比,YE1为基础的CBE系统(YE1-BE4)对ssDNA的催化效率降低了69倍之多。随后293T细胞中的ssDNA脱氨基实验也证实了以上结论。而全基因组测序分析也证实,YE1为基础的CBE系统的Cas9非依赖型DNA脱靶风险有明显降低,与单纯的nCas9风险相似(图1c-d)。
总体而言,研究者通过细菌中的利福平耐药和胸苷激酶基因突变实验以及人源细胞系中的R-loop实验、体外动力学实验和细胞内ssDNA脱氨基实验证实,较低的催化效率是CBE系统Cas9非依赖型DNA脱靶风险降低的基础。

图1 (a-b)R-loop实验分析不同CBE系统的Cas9非依赖型DNA脱靶风险;(c-d)全基因组测序分析YE1-BE4的DNA脱靶风险。
虽然YE1-BE4几无明显的Cas9非依赖型DNA脱靶风险,其活性窗口明显小于传统的CBE系统BE4,这限制了其应用。为此研究者尝试通过蛋白质工程拓展YE1为基础的CBE系统的活性窗口。首先,通过替换nCas9为nCas9-NG突变体,YE1-NG可识别的PAM位点从NGG拓宽为NG。研究者还通过替换nCas9为环化排列的Cas9(CP-Cas)变体CP1028(详见BioArt报道:NBT | David Liu又一力作:拓宽碱基编辑适用范围)得到YE1-BE4-CP1028,其活性窗口与BE4相似。随后为证实优化过的CBE系统在其它方面的安全性,研究者对YE1等rAPOBEC1突变体为基础的CBE系统的Cas9依赖型DNA脱靶风险进行检测,证实其Cas9依赖型DNA脱靶风险也明显降低。此外,YE1-BE3等多种CBE系统的RNA脱靶风险也明显降低。除蛋白质工程外,研究者还指出,通过核糖核蛋白转染等方式减少CBE系统在细胞内的存留时间也能有效降低Cas9非依赖型DNA脱靶风险。
总体而言,本研究通过一系列快速、便捷和经济的实验策略对多种CBE系统的Cas9非依赖型DNA脱靶风险进行了系统性的分析与比较,找到了降低Cas9非依赖型DNA脱靶风险的关键;并通过蛋白质工程等策略构建出一系列低脱靶风险的高活性CBE系统,为CBE系统的未来应用提供了助力。
值得一提的是,2月9日,中科院神经科学研究所杨辉团队在预印版平台bioRxiv发表文章High-fidelity base editor with no detectable genome-wide off-target effects。

David Liu文章先是在细菌(文章图1)和HEK293(文章图2)中建立了一套快速和成本更低的筛选Cas-independent脱靶的方法,发现YE1-BE4, YEE-BE4 and R33A + K34A-BE4等脱靶概率低。然后发现YE1-BE4的on-target效率更好(文章图3a,3b),并用WGS验证了YE1-BE4的脱靶效率确实是低。最后建立了YE1为基础的不同版本的base editors,发现YE1-BE4和BE4编辑效率差不多,但是DNA和RNA脱靶都很少。
杨辉团队的文章先是筛选了不同版本的base editors,挑选出on-target效率和WT base editor差不多的版本——YE1、FE1、R132E、R126E,然后用GOTI和RNA-seq分别检测他们的DNA 和RNA脱靶,发现YE1、FE1、R132E都不脱靶。
相同点:发现YE1是最好的版本,on-target效率不变,DNA和RNA脱靶同时减少;on-target编辑窗口都变窄;
不同点:David Liu是用多位点粗筛多方法快速找到脱靶低的candidate,然后用WGS验证。杨辉团队用GOTI和RNA-seq同时验证,发现了DNA脱靶和RNA脱靶并不完全一致,有些版本有DNA没有RNA脱靶,有些有RNA没有DNA脱靶,需要同时检测。
总体来说,这两篇文章用不同筛选策略,得到相似结果,很好的印证了彼此的发现。
原文链接:
https://doi.org/10.1038/s41587-020-0414-6
参考文献
1. Rees, H.A. & Liu, D.R. Base editing:precision chemistry on the genome and transcriptome of living cells. Nat Rev Genet (2018).
2. A. C. Komor, Y. B. Kim, M. S. Packer, J. A.Zuris, D. R. Liu, Programmable editing of a target base in genomic DNA withoutdouble-stranded DNA cleavage. Nature 533, 420-424 (2016).
3. Zuo E, Sun Y, Wei W, Yuan T, Ying W, Sun H,Yuan L, Steinmetz LM, Li Y, Yang H. Cytosine base editor generates substantialoff-target single-nucleotide variants in mouse embryos. Science. 2019 Apr19;364(6437):289-292.
4. Jin Shuai., Zong Yuan., Gao Qiang., ZhuZixu., Wang Yanpeng., Qin Peng., Liang Chengzhi., Wang Daowen., Qiu Jin-Long.,Zhang Feng., Gao Caixia., (2019). Cytosine, but not adenine, base editorsinduce genome-wide off-target mutations in rice., Science, 364, 292-295.
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652480805&idx=2&sn=bdc56316c06c271f3d7277104a8340f0&chksm=84e23691b395bf870aa37958c029d8d42c3bfd58fbdb102382b49c419e82b94c403d4d695bd7&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn



