近日,上海交通大学电子信息与电气工程学院自动化系潘小勇副教授团队在《Nature Communication》(自然-通讯)上以“ZeroBind: A protein-specific zero-shot predictor with subgraph matching for drug-target interactions”(基于子图匹配的蛋白特异性零样本药物靶标相互作用预测器)为题发表了最新的研究成果。该研究针对药物-靶蛋白相互作用预测在新药物和新蛋白表现不佳等方面的困难,提出了以特异性蛋白为单元使用元学习和弱监督的亚图信息瓶颈建模,在零样本和少样本场景中优于现有方法,弱监督地预测靶蛋白药物结合口袋。

研究背景
识别药物-靶标相互作用在药物研发中具有重要地位。通过计算机辅助药物设计、生物物理实验和高通量筛选等方法识别药物与靶标之间的相互作用,可以筛选出具有潜在疗效的化合物或药物候选物,更快地发现和开发新药物,降低研发成本,最终加速治疗方案的实施和改善病患的生活质量。
通过解析药物-蛋白质复合物的晶体结构来识别药物-靶标相互作用的传统实验方法,成本高昂且所需时间过长。与将大量候选药物直接纳入实验搜索相比,在实验搜索之前使用计算方法筛选出大多数候选药物更具有性价比。对接模拟利用药物分子和靶蛋白的3D结构来识别其潜在的结合位点,但依旧需要长时间的计算。相比之下,由于机器学习的快速发展,利用蛋白质和药物特征识别它们的相互作用具有低成本,高效率的优势。
创新成果
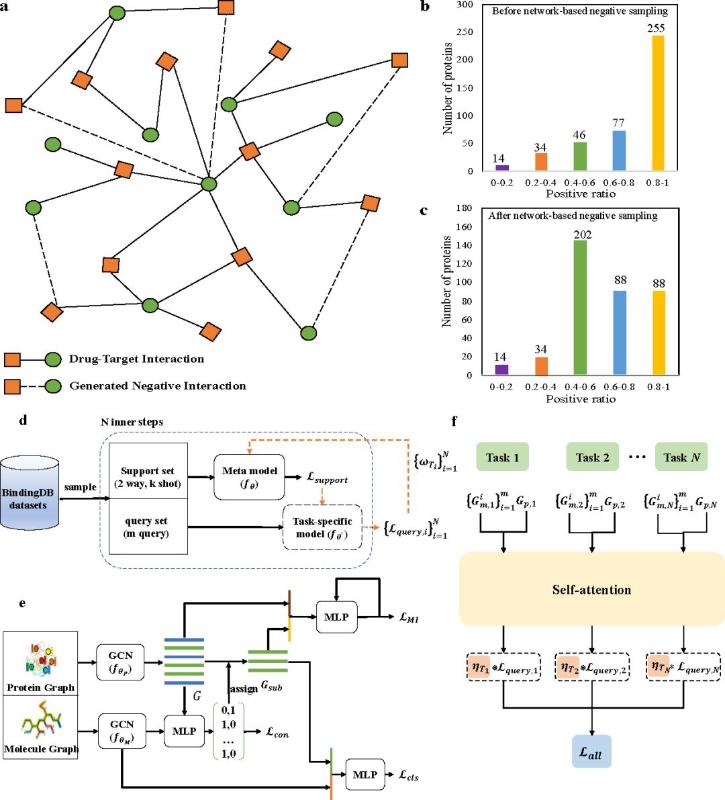
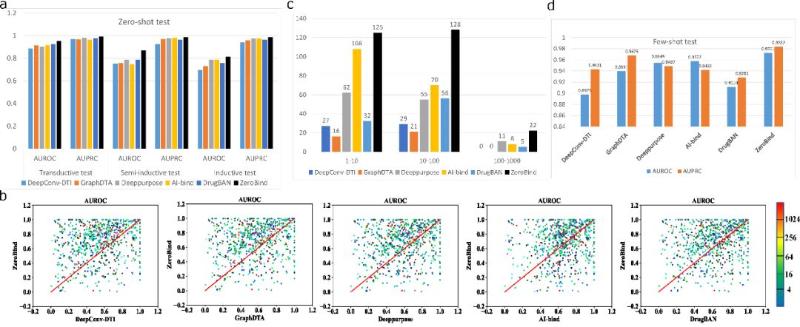
蛋白质特异性元学习框架——ZeroBind
论文提出了具有子图匹配功能的蛋白质特异性元学习框架——ZeroBind,将新药物和新蛋白质的药物-靶标相互作用预测作为零样本学习问题。首先,使用元学习策略从现有药物-靶标相互作用中学到的普适知识具有更强的泛化能力。其次,ZeroBind为每种蛋白质训练一个任务模型,每个特异性蛋白质模型将学习到该蛋白质特有的药物结合模式。论文还提出了与模型无关的最优子图学习,基于弱监督思想找到与该任务相关的最优子图作为蛋白质中的结合口袋,通过结合口袋特征进行相互作用预测,而不是使用包含大量冗余信息的所有蛋白质特征。ZeroBind在三个独立的零样本测试集和一个少样本测试集上进行了充分的实验,克服了现有方法的不足。模型学习的最优子图与蛋白质的真实结合口袋具有一致性,对药物-新冠蛋白靶点的结合预测效果也进一步验证了ZeroBind的可靠性。
论文信息
上海交通大学为论文第一完成单位,电院自动化系硕士生王宇轩为论文的第一作者,电院自动化系潘小勇副教授为通讯作者,电院博士生夏莹、严骏驰教授、袁野副教授和沈红斌教授为共同作者。该研究获得国家自然科学基金项目和科技部项目的资助。
论文链接:https://www.nature.com/articles/s41467-023-43597-1
作者:电子信息与电气工程学院供稿单位:电子信息与电气工程学院内容来源:上海交通大学
来源:上海交通大学
原文链接:https://news.sjtu.edu.cn//jdzh/20231218/192163.html
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn



