随着基因组测序技术的发展,物种和群体水平基因组数据呈指数增长。这些数据为在基因组水平鉴定和解析自然选择机制提供了机遇。然而,目前的分析方法面临着技术瓶颈和挑战。其中,关键问题是如何高效准确地检测作用于非编码区的自然选择效应。同时,能够高效、高性能地分析多物种大样本数据,成为方法学方面的迫切要求。
中国科学院北京基因组研究所(国家生物信息中心)陈华团队,在多物种联合等位基因频谱理论以及HKA(Hudson-Kreitman- Aguadé)检验的框架上,构建了CEGA(Comparative Evolutionary Genomic Analysis)方法。CEGA整合微进化过程与宏观进化过程模型,刻画了自然选择和群体历史在非编码区形成的遗传多态性“印记”,可高效、准确地检测作用于非编码区上的正向选择及平衡选择信号。CEGA同时分析物种间的分歧位点和物种内的多态位点信息。当两物种分化时间比较短时,多态位点蕴含的信息有助于准确推断分化时间、有效群体大小等信息,利于区分自然选择效应与群体历史干扰。因此,该方法在不同物种分化时间尺度上具有更广泛的适用性。仿真分析表明,对于不同的选择强度和物种分化时间,CEGA检测正选择与平衡选择的效果均优于现有方法,尤其对于选择强度较弱或者物种分化时间比较短的情景,CEGA的优势更为明显。除用于检测自然选择外,科研人员希望提出对自然选择发生过程的深入认知。 鉴于此,CEGA基于群体遗传学模型提出了关于自然选择强度等关键参数的推断。
该团队将CEGA应用于已发表的9个现代人类(Homo sapiens)和9个黑猩猩(Pan troglodytes ellioti)的群体基因组数据,进行编码区、非编码区两个层面上的比较分析,鉴定了在人类基因组中受自然选择作用而快速进化基因,并发现了这些基因的功能显著富集在与大脑容量、大脑皮层的总面积以及大脑皮层的厚度等相关表型和分子通路。此外,研究显示,在与免疫反应和病原体抵抗相关的区域(如主要组织相容性复合体MHC)存在显著的平衡选择信号。以上仿真分析以及人与黑猩猩基因组真实数据分析的结果表明,CEGA是有效的算法工具,可用于大规模群体基因组测序数据的高效分析。
相关研究成果以CEGA: a method for inferring natural selection by comparative population genomic analysis across species为题,发表在《基因组生物学》(Genome Biology)上。研究工作得到国家自然科学基金、国家重点研发计划和中国博士后科学基金等的支持。
论文链接
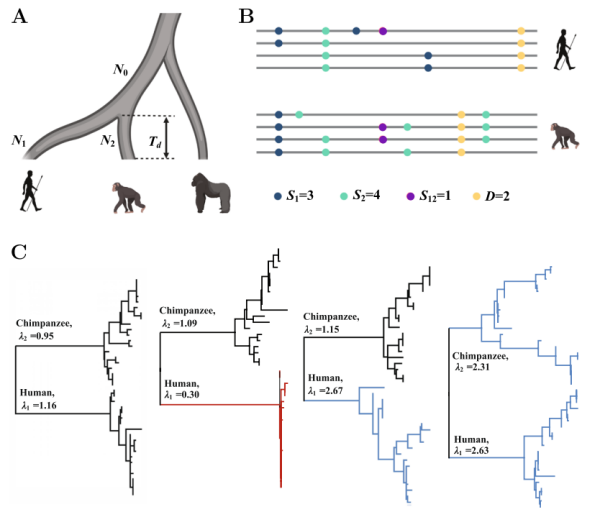
CEGA模型的参数及观测数据
内容来源:中国科学院
来源:中国科学院
原文链接:http://www.cas.cn/syky/202310/t20231017_4974851.shtml
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn