目前,CRISPR/Cas基因组编辑技术已成为功能基因组学研究和作物遗传改良的重要工具。但其编辑范围常受到PAM基序及其编辑窗口的限制。因此,开发具有更广泛PAM识别范围的Cas变体是扩大CRISPR/Cas系统编辑使用范围的一个重要研究方向。ScCas9是来源于狗链球菌(Streptococcus canis)的Cas9蛋白,与目前最常用的SpCas9蛋白(Streptococcus pyogenes, 识别NGG-PAM)具有89.2%的高度相似性,识别靶向范围更广的NNG-PAM,但其在动、植物中的编辑效率都较低,难以推广使用。前人通过对ScCas9的PAM-DNA互作结构域和loop基序中的氨基酸进行替换,获得了进化型的ScCas9+和ScCas9++变体,并在动物细胞中表现出高的编辑活性和广的编辑范围。然而ScCas9+和ScCas9++变体在植物中的编辑能力如何仍然不清楚。
JIPB近日在线发表了华南农业大学刘耀光院士/祝钦泷研究员团队题为“The ScCas9++ variant expands the CRISPR toolbox for genome editing in plants” 的研究论文(https://doi.org/10.1111/jipb.13164)。该研究利用来源于狗链球菌的优化的CRISPR/ScCas9++新变体,开发了广靶向的植物基因组编辑与胞嘧啶单碱基编辑器,为植物功能基因组学研究和作物遗传改良提供了新的研究工具。
该研究根据水稻密码子偏好性优化了ScCas9及其两个进化的ScCas9+和ScCas9++变体的编码DNA序列,系统地研究了它们对22个靶位点的编辑效率。结果发现ScCas9++的编辑效率比ScCas9和ScCas9+效率更高,但是它主要识别NGG-PAM靶点,而其它NNG靶点识别能力很低。该团队利用编辑效率最高的缺刻酶变体ScCas9n++构建了一个具有evoBE4max结构的新的胞苷碱基编辑器(CBE) PevoCDA1-ScCas9n++,在NNG-PAM上实现了稳定高效C−T的多重碱基编辑,并具有更宽的编辑窗口(C-1–C17),且没有靶点序列的偏好。进一步利用PevoCDA1-ScCas9n++对OsWx(3靶点)和OsEui1(2靶点)同时进行多靶点编辑,高效地产生了直链淀粉含量降低的新的优异水稻种质。该研究为ScCas9++变体在植物基因组编辑中的进一步开发与应用打下了基础、提供了思路。其缺刻酶变体ScCas9n++在碱基编辑器开发中具有更大的应用潜力。此CBE编辑器PevoCDA1-ScCas9n++为多靶点的单碱基编辑提供更为有效的工具,有望在基因功能筛选、大规模饱和突变、编辑调控元件、引入提前终止密码子或进行可变剪接等研究中有广泛应用。
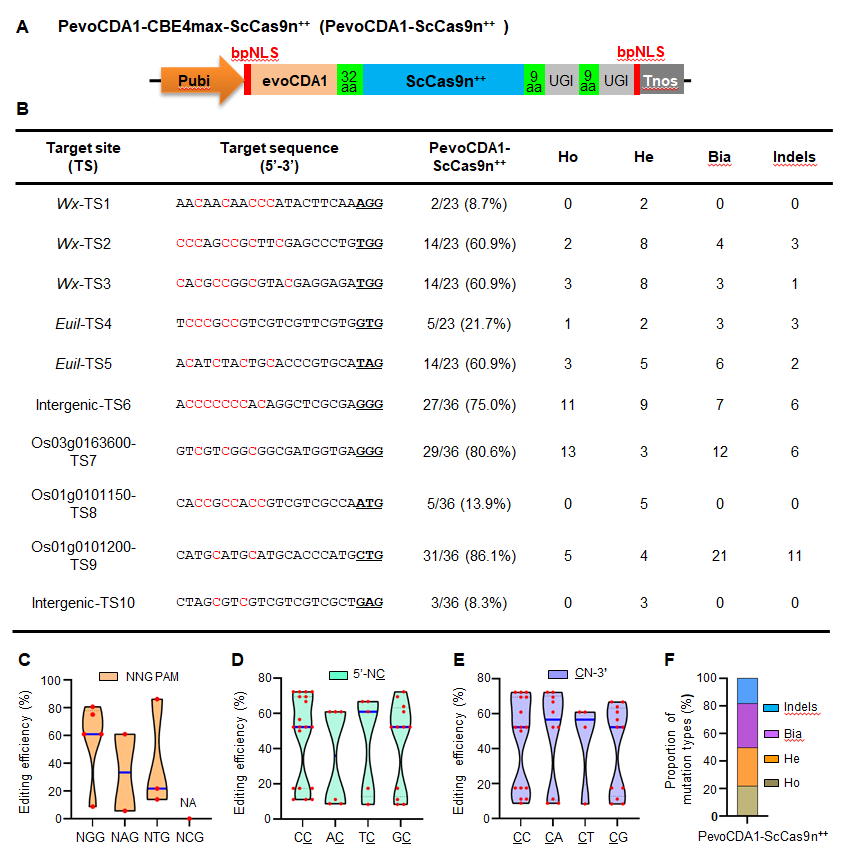
PevoCDA1-ScCas9n++的结构及其碱基编辑特性
刘耀光院士/祝钦泷研究员团队近年来在植物合成生物与基因组编辑技术开发的研究中取得了一系列进展,开发了多基因叠加系统TGSII,并创制了首个胚乳富含花青素与虾青素的“紫晶米”与“赤晶米”水稻新种质(Zhu et al., Mol Plant, 2017;Zhu et al., Mol Plant, 2018),同时还开发了广靶向基因组与单碱基的编辑系统(Zeng et al., Plant Biotechnol J, 2020a; Tan et al., Plant Biotechnol J, 2020; Zeng et al., Mol Plant, 2020),并创制直链淀粉含量适宜的品质优异的水稻新种质(Zeng et al., Plant Biotechnol J, 2020b)。这篇论文对进一步扩展植物基因组编辑工具箱的应用范围具有重要参考价值。硕士研究生刘涛利和博士后曾栋昌为论文的共同第一作者,刘耀光院士和祝钦泷研究员为论文的通讯作者。该研究得到广东省基础与应用基础研究重大项目、国家自然科学基金项目和广东特支计划科技创新青年拔尖人才专项的资助。
参考文献
Tan, J., Zhao, Y., Wang, B., Hao, Y., Wang, Y., Li, Y., Luo, W., Zong, W., Li, G., Chen, S., Ma, K., Xie, X., Chen, L., Liu, Y.-G., and Zhu Q. (2020). Efficient CRISPR/Cas9-based plant genomic fragment deletions by microhomology-mediated end joining. Plant Biotechnol. J. 18: 2161–2163.
Zeng, D., Li, X., Huang, J., Li, Y., Cai, S., Yu, W., Li, Y., Huang, Y., Xie, X., Gong, Q., Tan, J., Zheng, Z., Guo, M., Liu, Y.-G., and Zhu, Q. (2020a). Engineered Cas9 variant tools expand targeting scope of genome and base editing in rice. Plant Biotechnol. J. 18: 1348–1350.
Zeng, D., Liu, T., Ma, X., Wang, B., Zheng, Z., Zhang, Y., Xie, X., Yang, B., Zhao, Z., Zhu, Q., and Liu, Y.-G. (2020b).Quantitative regulation of Waxy expression by CRISPR/Cas9-based promoter and 5'UTR-intron editing improves grain quality in rice. Plant Biotechnol. J. 18: 2385–2387.
Zeng, D., Liu, T., Tan, J., Zhang, Y., Zheng, Z., Wang, B., Zhou, D., Xie, X., Guo, M., Liu, Y.-G., and Zhu, Q. (2020).PhieCBEs: Plant High-efficiency Cytidine Base Editors with Expanded Target Range. Mol. Plant 13: 1666–1669.
Zhu, Q., Yu, S., Zeng, D., Liu, H., Wang, H., Yang, Z., Xie, X., Shen, R., Tan, J., Li, H., Zhao, X., Zhang, Q., Chen, Y., Guo, J., Chen, L., and Liu, Y.-G. (2017). Development of “Purple Endosperm Rice” by engineering anthocyanin biosynthesis in the endosperm with a high-efficiency transgene stacking system. Mol. Plant 10: 918–929. (On the cover)
Zhu, Q., Zeng, D., Yu, S., Cui, C., Li, J., Li, H., Chen, J., Zhang, R., Zhao, X., Chen, L., and Liu, Y.-G. (2018). From Golden Rice to aSTARice: Bioengineering Astaxanthin Biosynthesis in Rice Endosperm. Mol. Plant 11: 1440–1448. (On the cover
来源:植物科学最前沿
原文链接:http://mp.weixin.qq.com/s?__biz=MzIyOTY2NDYyNQ==&mid=2247521873&idx=8&sn=857cd2a168819f9ba4ab9abe48343121
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn


