DNA甲基化是一种保守的表观遗传修饰,对基因表达和基因组稳定性具有重要意义。RNA介导的DNA甲基化(植物RdDM途径)是植物小RNA参与表观调控的重要方式,其需要两个植物特有的RNA聚合酶——Pol IV(大亚基NRPD1为催化核心)和Pol V(大亚基NRPE1为催化核心)以及大量的辅助蛋白。RdDM可以在转录水平抑制转座子和基因,并参与植物生物和非生物胁迫、植株再生、植物生长和果实成熟发育等生物学调控过程。
该研究中,研究人员通过全基因组甲基化测序以及差异甲基化区域(DMRs)分析发现,开花调控基因FVE能够影响部分RdDM通路调控位点的DNA甲基化水平。siRNA测序以及差异表达区域的分析说明FVE参与RdDM通路siRNA的调控,尤其是下游siRNA的累积。qRT-PCR实验分析说明FVE影响Pol V转录本的转录水平。此外,FVE调节DNA甲基化水平的变化伴随着相同趋势的siRNA水平的变化。染色体免疫沉淀测序试验(ChIP-seq)进一步说明了FVE倾向于结合RdDM通路下游调控位点,进而调节DNA甲基化。
虽然学界在RdDM途径和组蛋白修饰之间相互关系研究方面已取得深入进展,但现有的分子机制并不能完全阐述组蛋白修饰和RdDM作用位点的调控关系。中国科学院分子植物科学卓越创新中心研究员郎曌博课题组和南方科技大学杜嘉木研究组合作,通过正向遗传学筛选到了参与基因沉默的突变体,通过克隆发现是具有Tudor domain的基因RDM15功能缺失造成的。通过甲基化测序和DMR的分析发现,RDM15影响的甲基化位点是RdDM作用位点。该研究发现了一个新的H3K4me1识别蛋白RDM15,其通过招募Pol V来参与RNA介导的DNA甲基化过程的新机制。
通过FVE的免疫沉淀和质谱联用(IP-MS)IP-MS实验分析,研究人员发现FVE与RDM15蛋白能够相互作用,通过酵母双杂交实验(Y2H)和Split-LUC实验进一步确认了二者的互作关系。利用RDM15的全基因甲基化数据以及DMR分析,证明FVE和RDM15共同调节部分RdDM位点的DNA甲基化水平。FVE与RDM15的研究分析进一步确认了FVE参与RdDM通路下游功能调节的分子机制。
以上研究成果分别以MSI4/FVE is required for accumulation of 24-nt siRNAs and DNA methylation at a subset of target regions of RNA-directed DNA methylation和A histone H3K4me1-specific binding protein is required for siRNA accumulation and DNA methylation at a subset of loci targeted by RNA-directed DNA methylation为题,在线发表在The Plant Journal和Nature Communications上。
研究工作获得中科院战略性先导科技专项、国家自然科学基金等的资助。
论文链接:1、2
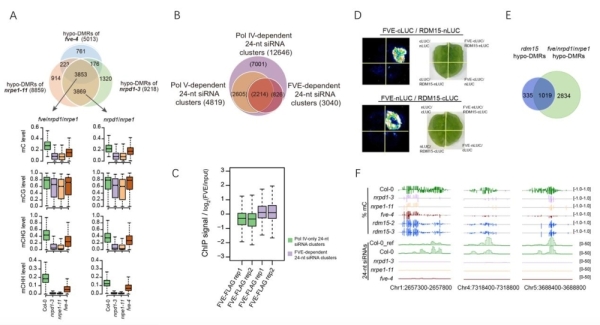
FVE与RDM15相互作用作用,调节RdDM通路相关位点的DNA甲基化和siRNA累积。FVE的DMR(A)、siRNA差异表达区域(B)以及染色质上结合位点(C)的分析实验,证实FVE参与RdDM通路下游调控位点的DNA甲基化和24-nt siRNA累积水平。Split-LUC等实验分析结果(D)验证FVE与RDM15相互作用,FVE与RDM15的DMR区域有很高的重叠(E),IGV示例(F)说明二者共同调节RdDM相关位点的DNA甲基化
内容来源:中国科学院原文链接:http://www.cas.cn/syky/202108/t20210803_4800974.shtml
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn