最近泛基因组研究揭示出一个物种内很大一部分基因区具有PAV变异。然而仅依赖编码区来评估物种水平上的功能基因组序列的变异并不完整。在泛基因组的研究中很少关注基因的非编码区域,即使这些序列能够直接调控基因的表达和表型。为了揭示非编码区域如何调节遗传变异,歇根州立大学Patrick P. Edger团队在Molecular biology and evolution 上发表了“Evolution of Conserved Noncoding Sequences in Arabidopsis thaliana”。该文章组装了分布于三十个栖息地的拟南芥基因组,其组装水平都达到了染色质级别,并且分析了保守非编码序列(CNS)在物种水平上的变化。文章揭示了CNS的PAV和位置变异 (PosV)也是非随机的。通过运用进化分析和利用染色可及性数据,作者进一步揭示了保守和可变的CNS在基因中的调控中的作用。同时作者分析还表明转座子元件有助于CNS变异。
保守的非编码DNA相比于蛋白编码基因而言依旧是一类未被充分研究的功能基因组特征。之前的植物比较基因组分析已经确定了一段长度通常为15-150个碱基对(bp)的非编码区域,在远缘物种中具有相同(或者接近相同)序列(fig. 1C)。这些序列普遍被认为是保守的非编码序列 (CNS)。之前的泛基因组研究都是研究转录区域存在PVA变异,以此来构建核心和泛基因区。作者认为泛CNS遵循泛基因的模式。尽管已经在植物基因组中鉴定了数以万计的CNS,但是这些比较通常是远亲物种单个样本间的比较。据作者所知,一个物种内多个个体基因组间CNS的变化规律并未在任何植物种揭示。因此作者组装了30种拟南芥基因组,并且利用最大的带注释的CNS数据集来研究CNS种内变异的水平和模式以及这种变异对于拟南芥基因表达的影响。
通常是通过比较不同物种间单个具有代表性个体的全基因组来鉴定CNS。因此这些序列在物种水平上的变异依然知之甚少,尤其是在植物中。作者研究了多个拟南芥种质中CNS结构的两种主要变异类型:PAV和PosV。他们定义了CNS的PAV变异是指在拟南芥生态型 (Col-0)中存在,但是在至少有一个其它的个体缺失的情况。PosV是指其它个体相对于Col-0而言其CNS的位置发生了变化(fig. 1A)。作者通过对十字花科中的九个系统发育信息类群进行了全基因组比对,得出了一组> 60000的CNS,其长度分布如fig. 1C所示。作者发现有209 (0.33%) 和951 (1.5%)个CNS表现出PAV和PosV。作者还发现他们的组装和长读段组装集之间存在明显的重叠 (fig. 1B)。这表明作者所利用的短读段的组装质量完全可以用于进一步分析。在30个短读长基因组组装分析所得出的62916个CNS中,作者发现有163 (0.26%),910 (1.4%) 分别表现出PAV和PosV (fig. 1D)。
为了分析CNS变异对于基因表达的影响,作者进一步研究CNS于其临近的基因之间的距离(fig. 2)。结果表明37.17% 的CNS � 500bp 位于基因的起始和终止位点。相对于在 Col-0 中保留其同线位置种质中的 CNS,PosV CNS 最近基因周围的 CNS 浓度有所降低。相对于邻近基因的位置似乎不会导致 CNS 表现出 PAV。先前的研究已经报道了染色质可及性与CNS具有重叠特征的有力证据。作者分析出的结果中有14%与之前的研究结果一致。他们展示了一个实例,用于说明CNS的丢失与染色质可及性的损失有关 (fig. 3)。作者结合RNA-seq数据用于分析CNS的缺失和获得与基因表达的直接关系 (table 1)。结果表明基因与CNS越相关,其在表达水平越高。反之表达水平越低。这说明了CNS与基因的表达水平显著相关。
此研究首次分析了植物种内水平上的CNS PAV和PosV。CNS序列在十字花科中表现出广泛的保守性,拟南芥中的可变CNS的比率虽然比可变基因组要小,但是比作者预期的更高。为什么近1000个PosV CNS位于不同的种质的不同位点上。作者给出了两种解释 (fig. 4)。首先作者提出了一个从头起源假说,作者发现PosV CNS的长度明显短于正常的CNS。通常要小20个碱基。因此可能大多数的CNS序列已经存在于可替代的基因座中了,仅仅需要对这些序列替换少量碱基即可将之转变为CNS (fig. 4A). 第二种假说是指调控元件的移动可能涉及转座元件 (fig. 4B)。
作者在这篇文章中证明了CNS的位置变异与染色质可及性显著相关。此外还证明了PosV CNS的功能,比如CNS的改变将会影响特异基因的表达,作者建议在拟南芥中应当进一步分析这一结果。这可能需要用到基因编辑技术来分析这些CNS的改变会对基因表达或是表型产生什么影响。作者还认为种群内的CNS变异能够作为衡量精华过程中的一种新机制。未来的研究还应当研究群体之间共享的CNS变异的比例的变化,以此来衡量选择性驱动过程是如何适应环境。
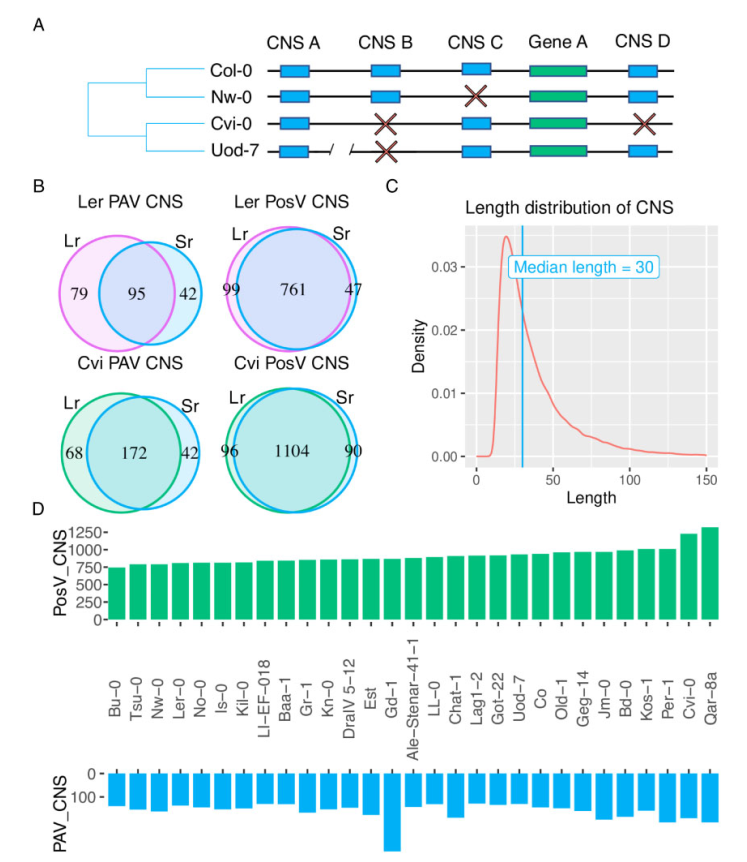
figure 1
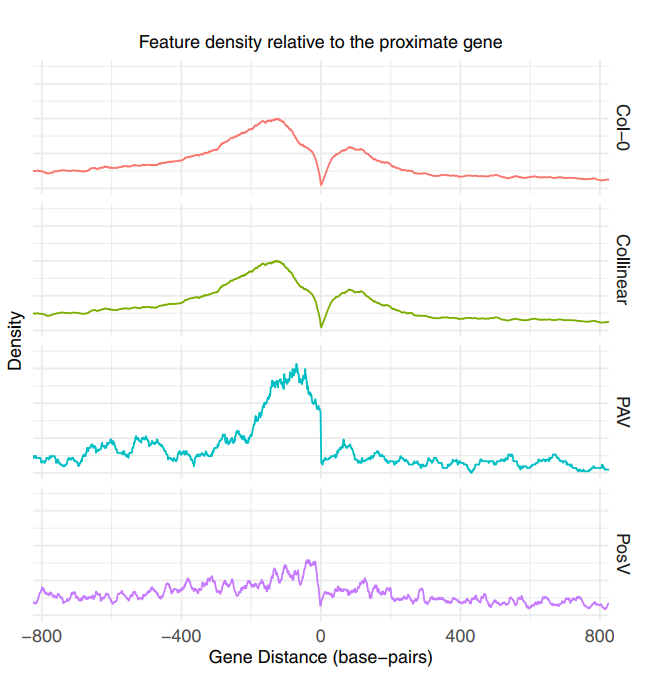
figure 2
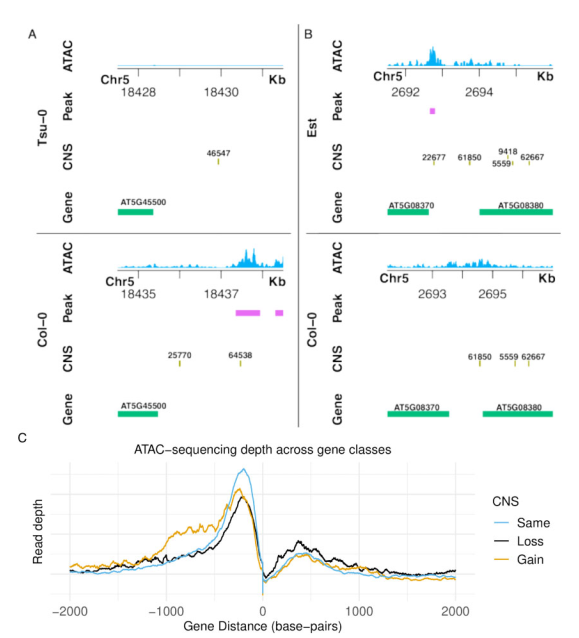
figure 3
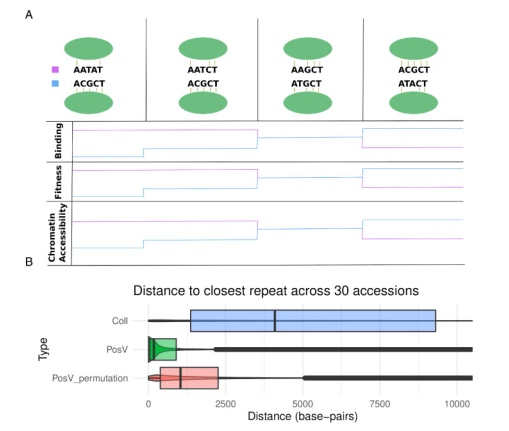
figure 4
来源:植物生物学
原文链接:http://mp.weixin.qq.com/s?__biz=MzI5NTk2MTcyOA==&mid=2247498382&idx=4&sn=5ecc067d8a77e0c7b5ed753ac5940d3d
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn