来源:BioArt
撰文 | 小柚
真核生物的染色质分为常染色质(通常为活跃转录区)和异染色质(通常为转录抑制区)。关于异染色质的形成和维持机制,近年来有多项研究从染色体结构和相分离的角度进行了阐述(详见BioArt报道:Molecular Cell | 复旦文波组发现异染色质稳定性维持的新机制;Molecular Cell | 李丕龙/李海涛合作报道组蛋白修饰通过促成相分离来调节染色质区室化的新机制;Nature | 核小体构象变化与异染色质相分离)。
除此之外,值得注意的是染色质的形成与空间分布密切相关。常染色质集中在细胞核的中间区域,而异染色质则分布在核周【1】。若将酵母或哺乳动物的一段基因固定到核周,则该基因表达也受到抑制。由此可见,核周分布对基因的沉默非常重要。组蛋白H3第9号赖氨酸的甲基化修饰,即H3K9me,是异染色质最重要的表观遗传标志。已有研究发现H3K9me的效应蛋白HP1可与核纤层蛋白Lamin结合【2】。但是核周分布是如何调控基因沉默和异染色质维持的,其中的机制尚不明确。
特别地,抑制基因转录的H3K9me可通过有丝分裂遗传,即在细胞分裂后被安置在子代细胞的相同基因区域;而对于激活型染色质区域来说,亲本核小体没有精确的分配到子代细胞相应的基因座位,而是随机分配的(详见BioArt报道:Cell | 染色质激活或抑制状态决定了核小体分离的差异性),异染色质的该遗传特性是否与其核周定位相关,有待进一步研究。
2019年12月26日,来自美国国立卫生研究院的Shiv I.S. Grewal教授在Cell发表研究Positioning Heterochromatin at the Nuclear Periphery Suppresses Histone Turnover to Promote Epigenetic Inheritance,该研究以裂殖酵母为模型,发现了Amo1蛋白通过结合RNA加工复合物RIXC和组蛋白伴侣复合物FACT介导异染色质的核周定位和遗传的分子机制。

在裂殖酵母中,mat2和mat3基因位于异染色质区域,边界明确,是研究异染色质功能的良好模型。
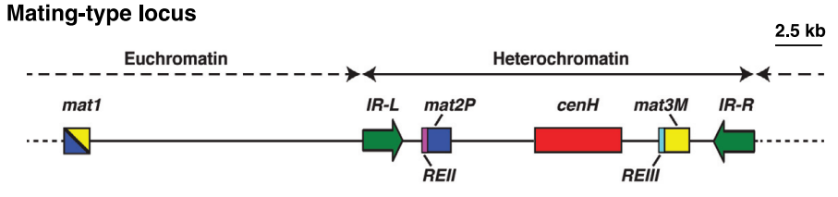
通过功能基因筛选,研究者首先确定了Amo1蛋白对该区域的异染色质维持非常重要。Amo1是一个非常保守的蛋白,在小鼠和人中均有同源蛋白。Amo1可能属于核孔复合物的一个部分,但也存在于非核孔区。研究者利用超高分辨率显微镜发现Amo1定位于核膜,并于核孔复合物有部分共定位,提示Amo1可能通过核孔复合物增强异染色质的沉默。
为了解析Amo1的作用机制,研究者利用蛋白免疫沉淀(IP)和质谱鉴定,发现Amo1与RNA加工复合物RIXC(由本研究作者命名,包括Rix在内的多个蛋白)以及组蛋白伴侣复合物FACT结合。
进一步分析显示,RIXC与Swi6(H3K9me效应蛋白HP1复合体成员)结合,使其分布在异染色质上,并包裹了异染色质的边界。Amo1与RIXC的结合,将异染色质带到核周围区域,这也就解释了为什么异染色质分布在核周。而异染色质的核周分布对FACT与Amo1和Swi6的结合至关重要。FACT可以维持异染色质基因沉默的状态,并抑制组蛋白的周转(turnover),实现异染色质在有丝分裂中的遗传。
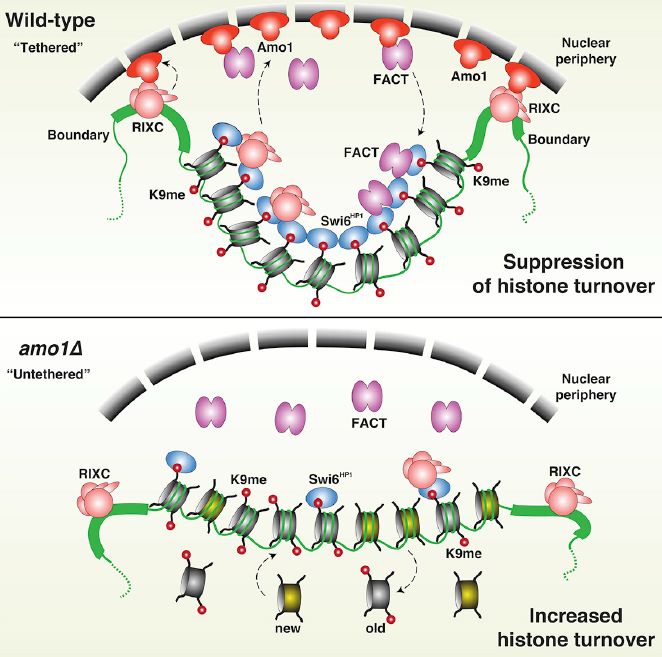
总的来说,该研究发现了异染色质在核周定位的保守机制以及该机制对异染色质基因沉默和遗传方式的重要性,具有重要意义。
值得注意的是,siRNA被报道可介导异染色质的形成【3】,而该研究鉴定的RIXC复合体可参与rRNA和tRNA的加工等过程,这是否与异染色质的形成,定位,维持和遗传相关呢?有待进一步的研究。
原文链接:
https://doi.org/10.1016/j.cell.2019.12.00
参考文献
1. Solovei, I., Kreysing, M., Lanctoˆ t, C., Ko¨ sem, S., Peichl, L., Cremer, T., Guck, J., and Joffe, B. (2009). Nuclear architecture of rod photoreceptor cells adapts to vision in mammalian evolution. Cell 137, 356–368.
2. van Steensel, B., and Belmont, A.S. (2017). Lamina-associated domains: links with chromosome architecture, heterochromatin, and gene repression. Cell 169, 780–791
3. Bayne, E.H., White, S.A., Kagansky, A., Bijos, D.A., Sanchez-Pulido, L., Hoe,K.L., Kim, D.U., Park, H.O., Ponting, C.P., Rappsilber, J., and Allshire, R.C.(2010). Stc1: a critical link between RNAi and chromatin modification required for heterochromatin integrity. Cell 140, 666–67
来源:BioGossip BioArt
原文链接:https://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652479456&idx=3&sn=d591805aed728dc30d3077ae9868f04a&chksm=84e20c54b39585424df039721218787e02e8a34bcafd23a8a807cee64364abd65ed67810bb6b#rd
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn



