来源:BioArt植物
近日,BMC Genomics 在线发表了中国农科院基因组所暨岭南现代农业科学与技术广东省实验室阮珏团队完成的题为 LRScaf: improving draft genomes using long noisy reads 的方法学论文。该方法应用于混合组装模式,不仅保证了组装结果的准确性和连续性,而且将运行时间降低了几个数量级,是大基因组混合组装的有力工具。

该研究基于混合组装策略,利用低深度的第三代测序技术超长读序,提高基于第二代测序技术的组装结果连续性。针对第三代测序技术的高碱基错误率所导致的大量假阳性比对,通过引入新的比对验证模型(如图 1 所示),有效地降低了其错误率的影响。基于String Graph的数据结构,以Contigs序列为顶点,长读序的连接信息为边,利用第三代测序技术超长读长的优势解决基因组中的复杂区域(如图 2 所示)。新研发的组装算法LRScaf (https://github.com/shingocat/lrscaf)能够高效和准确地完成基因组的组装。
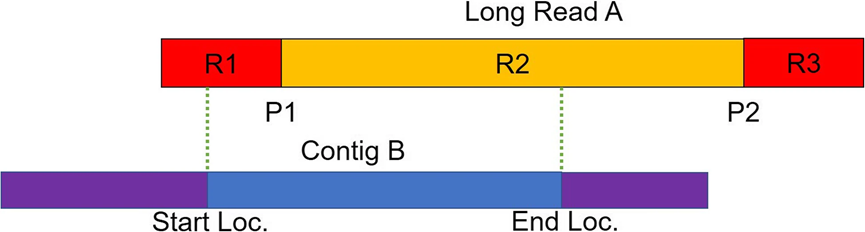
图 1 比对验证模型
在拟南芥和水稻的基准测试中,LRScaf的运行时间相较于SMIS降低了6700和4700倍,相较于MaSuRCA-Hybrid降低了1600和380倍。在人类的测试中,针对20x和35 x的PacBio和Nanopore原始长序列,本算法把CHM1的组装连续性(NG50)从127.5 kbp提升到10.4Mbp、把运行时间降低到1小时左右,内存使用峰值低于30.0GB;把NA12878的组装连续性从115.7 kbp提升到17.4 Mbp,把运行时间降低到2小时左右,内存使用峰值低于70.0 GB。
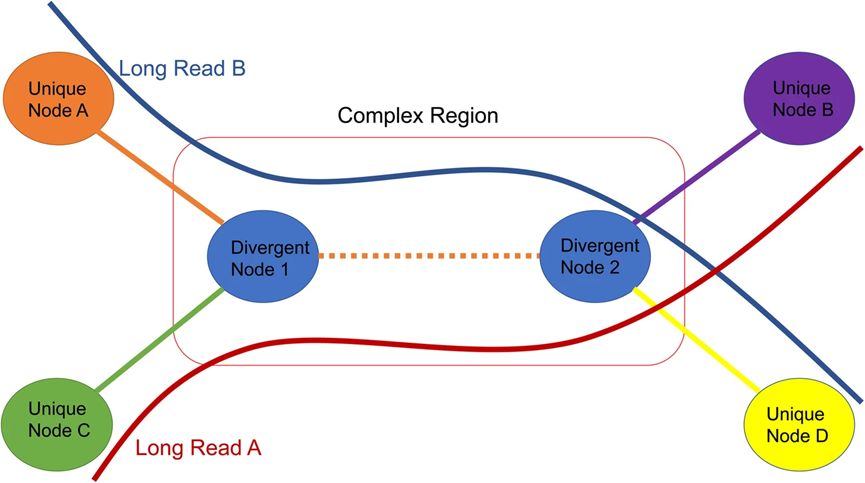
图 2 利用第三代测序技术超长读长的优势解决基因组复杂区域
该研究为大型基因组的混合组装提供了新的可行策略,为提升低连续性的组装结果和大型群体项目的组装提供了经济有效的方法,在基因组研究领域具有重要的意义。
中国农业科学院农业基因组研究所秦茂博士为论文的第一作者,阮珏研究员为通讯作者。该研究得到了国家自然科学基金面上项目和深圳大鹏新区产业发展专项资金等项目的资助。
来源:基因组所。
文章链接:
https://bmcgenomics.biomedcentral.com/articles/10.1186/s12864-019-6337-2
来源:bioartplants BioArt植物
原文链接:http://mp.weixin.qq.com/s?__biz=MzU3ODY3MDM0NA==&mid=2247493285&idx=5&sn=ef971bb9de58597ba386cbfbe38d2174&chksm=fd737cc2ca04f5d43ac1cf94736d532ad12cceb786237cd1656013283f46ad9e154e17bfc989&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn



