来源:BioArt
撰文 | 章台柳
肿瘤生物学领域的一个前沿研究是利用大规模细胞测序分析肿瘤细胞群内的分子动态变化。然而,目前来说,单细胞基因组测序技术由于捕获效率问题要落后于转录组学【1】。但是,通过对组织和细胞群进行单细胞基因组分析,将会极大地促进对诸如恶性组织的克隆组成解析(clonal decomposition,也就是克隆异质性),罕见细胞群的基因型鉴定、单个细胞的DNA复制状态识别等的研究,而这些信息则无法通过多细胞测序(bulk sequencing)所识。目前存在几种以扩增为基础的方法,如简并核苷酸引物PCR(DOP-PCR)、多重置换扩增(MDA)、多重退火-环状循环扩增(MALBAC),但是扩增过程存在的覆盖率和聚合酶偏好等问题,导致得到的数据有一定的失真,同时重复序列也较难分析。近期出现的单细胞组合标记测序技术(SCI-seq)能够提高单细胞测序的规模,但也存在重复率高和覆盖度低的问题【2】。
2017年,加拿大BC癌症研究中心Samuel Aparicio团队在Nature Methods杂志上曾发表过DLP方法(direct library preparation)【3】,即用微流控设备获得单细胞并裂解,在微小体积(纳升nl)内直接加入标签序列并打断基因组,随后通过PCR加入索引条形码(index barcodes)和测序接头(sequencing adaptors),完成单细胞文库的制备,该技术能够减少基于预扩增技术的偏差。但是微流控设备限制了实验的规模,对细胞的大小也有要求,大细胞堵塞通道,小细胞无法捕捉。基于微滴的单细胞测序对细胞的大小也有类似的限制。因此单细胞测序技术需要进一步优化。
2019年11月14日,Samuel Aparicio 和Sohrab P. Shah合作在Cell杂志上发表文章Clonal Decomposition and DNA Replication States Defined by Scaled Single-Cell Genome Sequencing,开发了一个新型高通量单细胞基因组测序平台——DLP+。利用DLP+平台对包括细胞系、异种移植物和临床样本等共51926个单细胞进行单细胞基因组测序并记录了相应的细胞图像。DLP+的数据可以用来鉴定不同组织类型和基因型间有丝分裂错误分离率的变化、细胞形态和基因组倍性状态之间的相关性;利用共享拷贝数信息对细胞进行聚类,计算了单核苷酸分辨率的克隆基因型,推断克隆的系统发育;细胞群体中克隆特异性染色体的非整倍体特性。

为了将DLP平台建库的规模扩增到上千个单细胞,研究人员利用现有的商品组件搭建了新型平台DLP+,主要有非接触式压电分配器、开放式纳米孔阵列组成(实现各种大小的单细胞的获取),可实现大量自由编程(可灵活调整文库建立的方式)、实时成像和对象识别(新增图像信息)等功能。实时成像有效排除双细胞、空细胞或受污染的细胞的情况。最终文库汇总,利用HiSeq平台进行测序。研究人员开发了开源的云兼容的软件系统,便于DLP+平台的数据分析。
利用DLP+平台,研究团队对多种组织和细胞进行测序,包括细胞系、人乳腺癌来源的异种移植物、小鼠滑膜肉瘤、滤泡性淋巴瘤患者的冷冻样本、乳腺癌患者的穿刺诊断样本和快速冷冻组织中的细胞核等,构建了高质量的注释单细胞基因组的资源库。DLP+不仅可以在建库过程中利用自发荧光将死细胞选择性剔除,而且可以利用测序质量评判区分细胞活性。荧光判定为活细胞中,65%可以产生高质量的单细胞基因组测序数据;每个样本中,活细胞的成功率在30.6%~96.0%,中位数为73.3%,而28/33的样本中活细胞成功率超过50%。死细胞的质量分数远低于活细胞。细胞核文库产生高质量数据的成功率为66.0%,其质量分数、总比对读值、重复读值和整合度等质量指标都与细胞文库制备类似。高质量的文库可以用于识别高度非整倍体的基因组状态,包括复杂的重排。
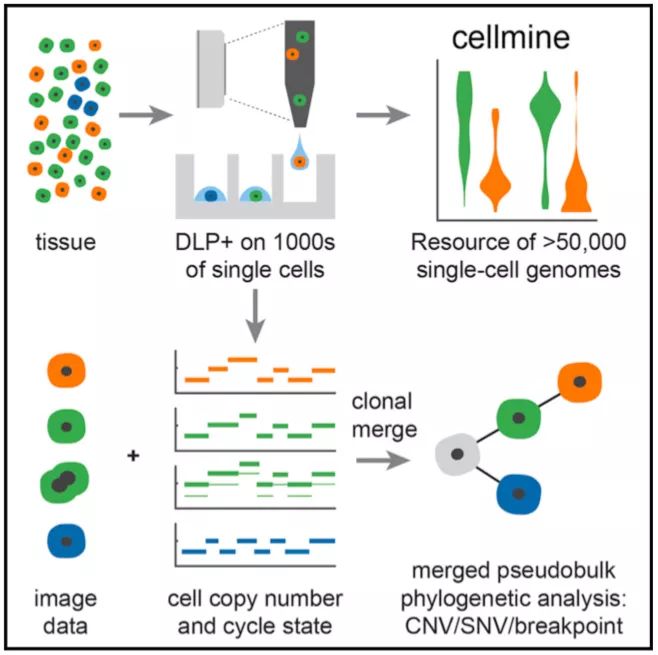
利用DLP+平台对大量单细胞基因组进行低覆盖率的测序,可以用于确定克隆群体,进而推断克隆特异的体细胞突变事件如SNVs、等位基因特异性拷贝数、重排断点,并对系统发生进行计算。对3个克隆相关的晚期血清型卵巢癌细胞系——来源于同一病人的原发肿瘤和2个复发肿瘤样本的1966个细胞进行DLP+建库。根据共享拷贝数鉴定出9个细胞亚群,并计算克隆特异性特征如总克隆数、等位基因特异性拷贝数、SNVs和断点。例如,克隆E的总拷贝数和次要等位基因片段大体上与全基因组复制事件相一致。染色体1、7、10、11有4个拷贝,次要等位基因片段接近0.5;染色体2、5、9包含3个拷贝的片段,次要等位基因片段为0.33,表现出单拷贝数丢失;染色体3、4、6、12有5个拷贝的片段,次要等位基因片段为0.4,表现数单拷贝增加。此外,DLP+可以分辨出克隆特异的局部扩增,如克隆1,8的13号染色体的4克隆片段。SNVs分析显示3个样本在系统发育中都形成了不同的分支。在14068个SNVs中,84%与推测的系统发生树完全吻合,其中28%预测为祖细胞相关如TP53、FOXP2、SUGCT,9%克隆特异,其余63%为分支特异性如ZHX1、HTR1D、INSL4。对断点进行分析,发现15%预测为祖细胞相关,11%为克隆特异,73%为分支特异性,与SNV的系统发生分类特征类似。
单细胞基因组中,整条染色体的非克隆性增加和丢失可用于观测有丝分裂的错误分离。对未分选过的二倍体细胞的DLP+文库进行分析发现,少数细胞(少于5%)具有大体的二倍体基因组,但具有一个或多个完整染色体的非整数倍,即染色体分离错误。不同细胞类型的常染色体有丝分裂错误率不同,如184-hTERT野生型和p53缺失细胞系中为5.2%,而参考GM18507细胞系的比率为2.6%。同时,对于淋巴细胞型和乳腺上皮型184-hTERT细胞,完整染色体的获得比丢失更占优势。而且,虽然184-hTERT野生型和p53缺失细胞系的完整染色体非整数倍发生的比率类似,但是p53缺失型中完整染色体的丢失高于获得。3种细胞类型中,22个常染色体的获得和丢失具有相似的数量级,与染色体的大小无关,也没有染色体特异性。
DLP+单细胞基因组测序可以用来检测DNA复制的中间状态。人类细胞中,基因组的复制具有非同步性的特征,早期、晚期复制的区域其GC含量不同。处于S期的细胞有强烈的GC偏好,在G1或G2中没有。经过对GC的适当矫正后,S期细胞表现出部分复制的拷贝数特征,其中早期复制区域的拷贝数状态高于晚期复制区域。对正在细胞周期内的T-47D人乳腺癌细胞系进行DLP+建库分析,发现单细胞基因组的深度测序能够清楚地识别出罕见的染色体非整倍体状态,这种非整倍体状态在增殖和复制过程中不会扩增。
最后,研究人员对细胞形态和基因组特征之间的关系进行研究。使用6个乳腺癌异种移植物的单细胞基因组测序数据和匹配的细胞图像,发现从G1期到G2期细胞直径显著增加。酵母的研究显示S期的核大小并没有显著增加,提示核大小不仅仅决定于DNA的含量。但是,研究发现乳腺癌异种移植物的整倍体增加与细胞、细胞核的大小相关。
总的来说,研究开发了一种新型单细胞基因组测序的流程方法,不仅规避了扩增带来的弊端,提高了规模化,而且保留了细胞或细胞核的图像资料;同时展示了如何利用单细胞基因组测序数据鉴定克隆群体及其基因组特征、单个细胞的特征(复制状态和染色体错误分离)以及基因组核形态学特征之间的关系,为研究单细胞的基因组提供了新的工具和资源库。
原文链接:
https://doi.org/10.1016/j.cell.2019.10.026
参考文献
1. Gawad, C., Koh, W., and Quake, S.R. (2016). Single-cell genome sequencing: current state of the science. Nat. Rev. Genet. 17, 175–188.
2. Vitak, S.A., Torkenczy, K.A., Rosenkrantz, J.L., Fields, A.J., Christiansen, L., Wong, M.H., Carbone, L., Steemers, F.J., and Adey, A. (2017). Sequencing thousands of single-cell genomes with combinatorial indexing. Nat. Methods 14, 302–308.
3. Zahn, H., Steif, A., Laks, E., Eirew, P., VanInsberghe, M., Shah, S.P., Aparicio, S., and Hansen, C.L. (2017). Scalable whole-genome single-cell library prepa- ration without preamplification. Nat. Methods 14, 167–173.
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652478634&idx=3&sn=272cfc949bf76dc260a0f4c7d9e9bcee&chksm=84e20f1eb39586082dad5ca12e587ea5c3116c88856451de34bb9395d915e2470bd56d855252&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn


