1. 摘要:
死亡微生物的胞外DNA可以在土壤中保存数周至数年。虽然人们隐式地认为从土壤中提取的微生物DNA主要代表完整的细胞,但目前还不清楚细胞外DNA如何影响微生物多样性的分子分析。我们使用基于光活性DNA涂层染料叠氮丙啶的活性PCR检测了广泛的土壤。我们发现,平均而言,40%的原核和真菌DNA都是细胞外的或来自不再完整的细胞。细胞外DNA使所观察到的原核和真菌丰富度增加了55%,并导致对分类单元相对丰富度的严重误估,包括对关键生态系统过程不可或缺的分类单元的相对丰富度。细胞外DNA在所有土壤中均不存在,在低交换性碱基阳离子浓度的土壤中更易出现,在高pH土壤中,其去除对微生物群落结构的影响更为深远。总之,这些发现表明,细胞死亡后残留在土壤中的“残余DNA”可能会模糊处理效果、时空模式以及微生物类群与环境条件之间的关系。
2. 研究进展:
(1)先前的研究表明,细胞死亡后,可扩增的细胞外DNA可在土壤中持续数周至数年。(Nielsen et al., 2007; Pietramellara et al., 2009; Levy-Booth et al., 2007)。(2)细胞外DNA库的寿命和大小由无数复杂的物理和生物因素控制。例如,土壤矿物学、pH值和离子强度可以控制DNA对土壤基质的吸附,以及DNA本身的分子完整性。(Ogram et al., 1988; Saeki et al,. 2010)
(3)吸附的细胞外DNA降低了微生物的可及性,从而限制了其通过合成代谢或分解代谢过程或作为可转化遗传物质来源从环境中去除。(Nielsen et al,. 2006)
3. 提出假设:
考虑到细胞外DNA在土壤中存在的可能性,我们假设细胞外DNA可能会影响基于DNA的土壤微生物群落多样性和结构的估计。在这里,我们将“残留DNA”定义为细胞外或细胞内不再完整的DNA,从而被光活性活性PCR指示剂单叠氮丙啶(PMA)去除。
4. 材料与方法:
2015年8月到9月,共从美国收集了31种表土(0-5cm,仅矿物土)。来自外地的样品被运送,冷却,过夜。重要的是,为了防止潜在的冷冻对细胞膜的损伤,在PMA处理之前,土壤故意不被冷冻。到达实验室后,将土壤筛至2.0 mm,均匀化并在4°C下储存,直到PMA处理。每种土壤的子样本要么用PMA处理(n=5),要么不处理(n=5)。
PMA是一种具有光活性的DNA嵌入染料,通常被膜完整的细胞所排斥,但与残留DNA(胞外DNA和胞质膜受损细胞的DNA)结合。当暴露在光下时,插层PMA永久性地修饰DNA,使其不能被PCR扩增。
使用qPCR计算残留DNA的数量,方法是从PMA处理的样本(来自完整细胞的DNA)中的可扩增原核16srrna基因或真菌内转录间隔区1(ITS)扩增子的丰度减去未处理样本(总DNA=来自完整细胞的DNA+残留DNA)中的这些扩增子的丰度。通过对PMA处理和未处理土壤亚样品中扩增的rRNA基因区域(原核生物为16srrna,真菌为ITS区域)的高通量测序,对微生物群落进行了表征。在将所有文库标准化到同等测序深度后,我们比较了微生物丰富度、总体群落组成和单个分类群相对丰富度的估计值。
5. 结果:
(1)残留DNA代表了土壤中微生物DNA的很大一部分。在所有31种土壤中,可扩增的原核16S rRNA基因的40.7±3.75%(平均值±se;n=155)是从残余DNA库中获得的。在真菌中也观察到类似的模式,其中40.5±4.12%(平均值±se;n=155)的真菌其扩增子来源于残余DNA池。去除残留DNA显著降低了对土壤微生物多样性的估计。在所有样本中,去除残留DNA后,原核生物的总丰富度降低了13.9±1.20%(平均值±se;n=155)。去除残余DNA后,真菌丰富度平均下降了12.4±1.97%(平均值±se;n=152)。
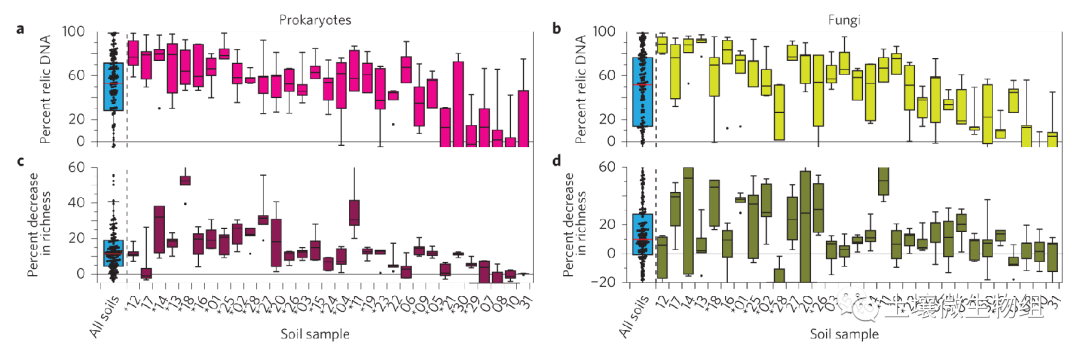
Figure 1. Relic DNA inflates soil microbial diversity estimates. a,b, Percent of total prokaryotic 16S rRNA gene copies (a) or fungal ITS copies (b)inthe relic DNA pool, as quantified by qPCR. c,d, Percent decrease in total prokaryotic (c) or fungal (d) richness after removal of relic DNA, as estimated from rarefied amplicon sequence data.
(2)就单个土壤而言,在所有31种被测土壤中,原核生物群落的组成都受到残留DNA去除的显著影响(PERMANOVA R2=0.10-0.23,q值≤0.05)(图2a)。在31种土壤中,有21种土壤中残留DNA的去除对真菌群落的组成也有显著影响(PERMANOVA R2=0.10-0.22,q值≤0.05)(图2b)。
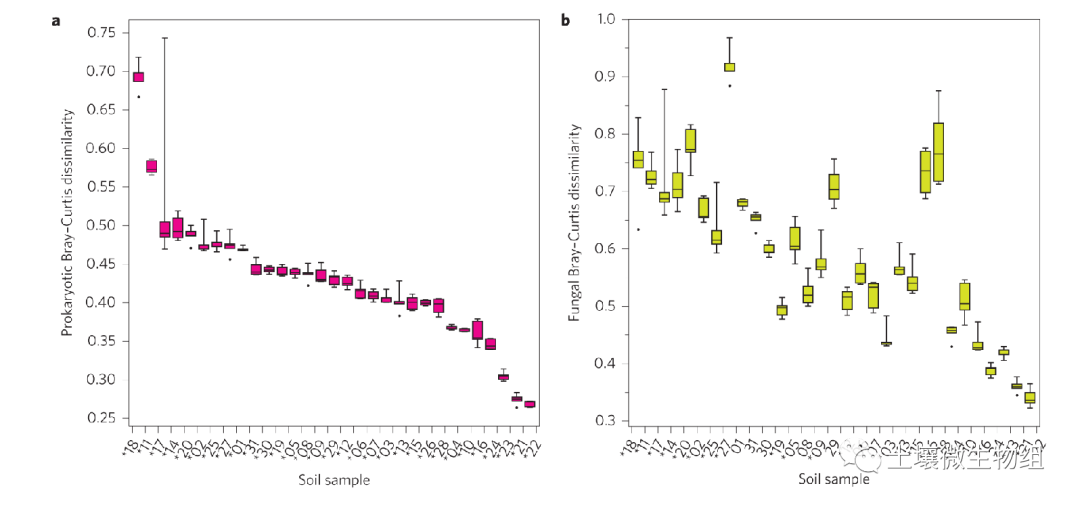
Figure 2. Relic DNA removalcan haveasignificant effect on community structure within a given soil sample. a,b, Mean dissimilarity in soil prokaryotic (a) and fungal (b) communities after relic DNA removal, relative to untreated soils.
(3)去除残留DNA后,土壤中大量微生物的相对丰度发生了变化,但变化的类群和观察到的移动方向不同。例如,在美国东北部的一个草原土壤(土壤25),放线菌和α-变形菌在去除残余DNA后相对丰度显著增加,但疣微菌减少(图3)。在许多情况下,残留DNA去除后估计的相对丰度变化接近或超过25%(图3)。
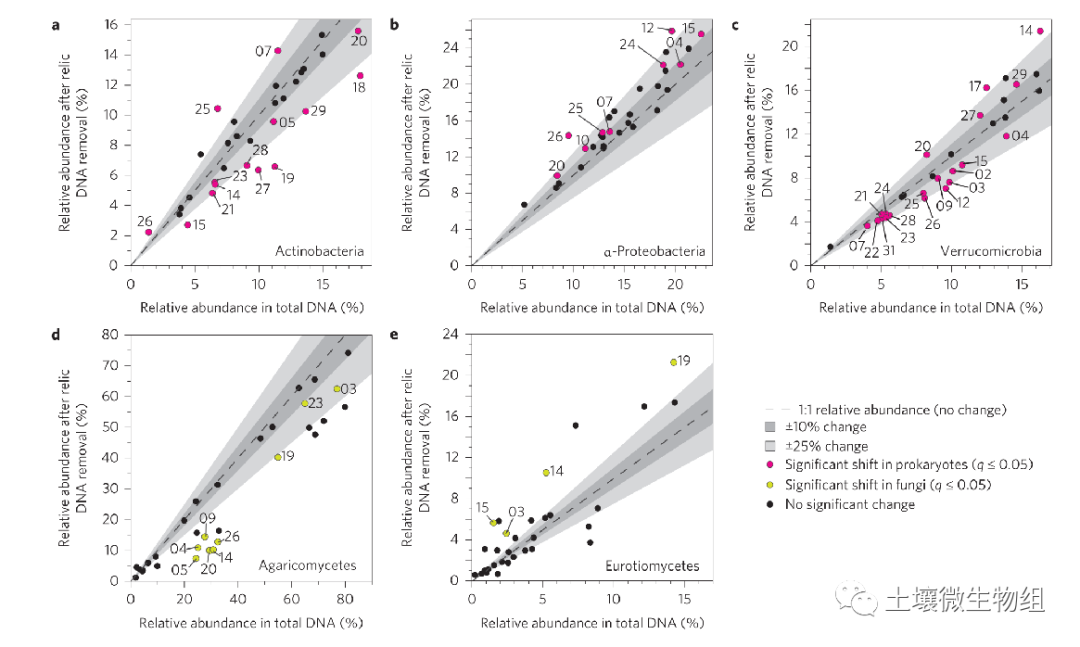
Figure 3. The relative abundances of several microbial lineages change significantly in individual soils after removal of relic DNA.
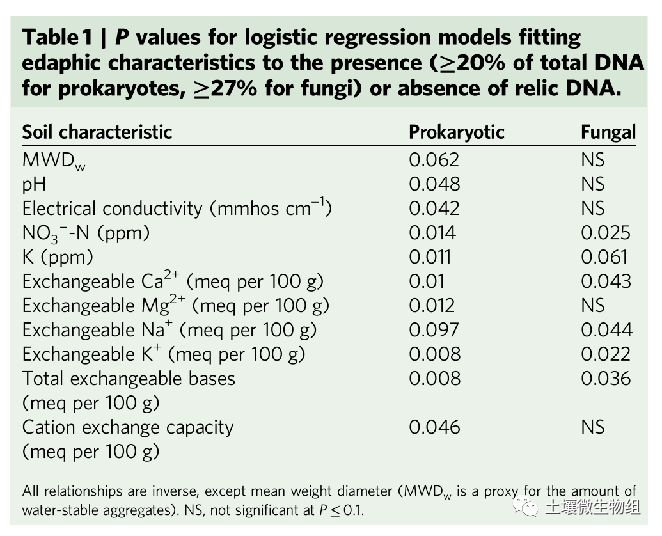
(4)我们发现已知影响DNA和土壤颗粒之间静电相互作用的土壤特征是微生物残余DNA存在的显著预测因子。例如,具有少量可交换碱基的土壤,特别是K+,可能含有来自原核和真菌来源的残余DNA。尽管低pH值、低电导率和低阳离子交换容量的土壤更有可能含有残余DNA,但原核生物的这种倾向比真菌的强。
6. 总结和讨论:
我们发现,残留DNA可导致土壤微生物多样性的显著高估,并降低准确定量原核和真菌群落结构的能力,这具有若干重要意义。
首先,它表明土壤中的实际微生物多样性比经常报告的要低。
第二,尽管残留DNA的存在不太可能掩盖广泛的生物地理学模式,但残留DNA可能掩盖土壤条件实验操作中微妙的时空模式或处理效果。例如,土壤微生物群落在不同季节的变化,或随着植物物种在某一地点的生长,通常很难从基于DNA的分析中检测出来。同样,研究气候变化对土壤微生物群落影响的长期土壤移植试验表明,微生物群落组成的变化很小。我们预计,如果通过首先去除残留DNA来减少非生物微生物产生的“噪音”,那么检测土壤微生物群落有意义变化的能力应该会提高。
最后,土壤微生物群落的极端多样性为亚基因组组装和分析提出了多重计算问题。由于这种多样性的很大一部分来自残余DNA库,因此在鸟枪元基因组分析之前从样品中去除残余DNA应能产生更有效的元基因组组合,并提高我们推断未记录土壤微生物基因组特性的能力。残留DNA动态可能对理解土壤以外其他生态系统中的微生物群落有重要影响。例如,深海沉积物中含有大量的胞外DNA,这表明去除残余DNA可能会影响深层生物圈的多样性估计。对深层生物圈样本中的残留DNA库进行具体分析,作为“化石记录”可能具有特殊的用途,以区分已灭绝的微生物类群和活生物体,并更准确地重建地下古海洋微生物群落。更普遍地说,残留DNA可能会影响死亡生物体DNA含量丰度或DNA特别耐降解的研究,包括对水生颗粒、矿物表面或建筑环境中发现的微生物的研究。
7. 参考文献:
Paul, Carini, Patrick, et al. Relic DNA is abundant in soil and obscures estimates of soil microbial diversity.[J]. Nature microbiology, 2016.
来源:meta-genome 宏基因组
原文链接:http://mp.weixin.qq.com/s?__biz=MzUzMjA4Njc1MA==&mid=2247499016&idx=4&sn=7640be19e0b63e9714101760c86b26b7
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn