来源:植物科学最前沿
近年来,随着测序技术、组装方法和计算资源的改进,作物基因组学取得了巨大的进展,也促进了作物改良新工具的开发。其中,物种内结构变异和泛基因组特征的研究揭示了物种内个体间广泛的基因组差异,为农作物基因组学研究和遗传改良提供重要基础。
2021年1月4日,国际知名杂志Genome Biology在线发表了美国爱荷华州立大学生态、进化与有机生物学系的Matthew B. Hufford和明尼苏达大学农学和植物遗传学系的Candice N. Hirsch教授的综述论文《How the pan-genome is changing crop genomics and improvement》,回顾了作物基因组学和泛基因组学的研究进展。 20世纪初,植物基因组测序彻底改变了作物生物学和育种的思路。早期的组装使我们能够在SNPs水平更深入地了解植物物种的多样性。之后,广泛的结构变异(SV)(PAV,CNV和染色体重排)被鉴定到。在物种内,基因组在基因含量(如串联重复基因、散布在基因组中的CNV和基因的PAVs)和基因组的重复序列比例(如转座因子、knob repeats和着丝粒重复)上都存在差异的现象。于是“core”基因组和“dispensable”基因组的概念提出用于描述泛基因组中普遍存在的这种变异。
20世纪初,植物基因组测序彻底改变了作物生物学和育种的思路。早期的组装使我们能够在SNPs水平更深入地了解植物物种的多样性。之后,广泛的结构变异(SV)(PAV,CNV和染色体重排)被鉴定到。在物种内,基因组在基因含量(如串联重复基因、散布在基因组中的CNV和基因的PAVs)和基因组的重复序列比例(如转座因子、knob repeats和着丝粒重复)上都存在差异的现象。于是“core”基因组和“dispensable”基因组的概念提出用于描述泛基因组中普遍存在的这种变异。
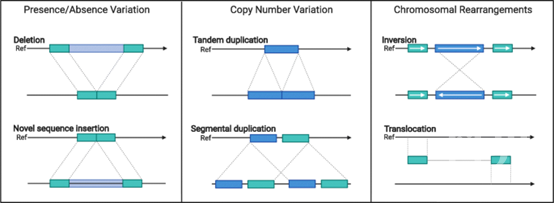
随着对泛基因组结构变异的深入理解,我们对作物基因组学的思考方式正在改变。早期对所研究性状的遗传结构(QTL和GWAS)以及基因组预测主要依赖于SNP标记。在泛基因组时代发现的结构变异需要重新评估其对表型的决定因素。迄今为止,已有报道发现结构变异与环境适应有关,如对非生物和生物胁迫的耐受性和开花时间;此外,植物的驯化特性,如不破碎和植物株型的变化均是由SVs引起的。考虑到SV对植物性状的重要影响,它们的特征对于作物的驯化和改良有着重要的意义。
组装方法和生物信息学的提升使得作物泛基因组的特征化成为可能
作物基因组组装技术的改进
最近,三代测序技术的成熟促进了作物基因组可获得更为连续和完整的组装。某些物种中存在单个物种内的多个基于long-read的组装。除其他重要发现外,这些不同的组装也促进了非编码和调控变异与农艺性状间相关性的发现。
基于单个参考基因组的结构变异特征
随着Illunima重测序成本的降低和普及,全基因组测序(WGS)方法广泛的地用挖掘作物中存在的CNV/PAV。但这种方法也有一定的局限性:(1)参考基因组的不完全组装或真正的个体间的缺失使得某些基因座序列不能很好的被鉴定;(2)不同的reads map效率较低,(3)短读取序列的不均匀覆盖偏差可能导致不准确的鉴定。
利用最近开发的文库制备技术和成熟的单分子长reads测序技术,如10x,Hi-C和Strand-Seq可以通过构建特殊的文库,使用短的reads来获取较长的基因组信息。Bionano,PacBio和Oxford Nanopore等测序技术能够获得参考基因组中缺失的复杂序列。这些技术的创新导致了迄今为止最全面的CNV/PAV基因组信息的鉴定。然而,测序成本仍限制其在作物中的广泛使用。
通过构建泛基因组来鉴定结构变异
在一个物种内获得多个高质量参考基因组提供了以非参考偏差的方式进行SV鉴定的机会。然而,一些作物物种庞大而复杂的基因组特征,使得每个taxon物种的大量组装成本令人望而却步。单倍型分离在一定程度上可以降低这一局限性;其次,虽然多个组装能够减少参考偏差,但组装错误可能会导致检测到错误的SV并影响下游分析。最后将泛基因组变异整合到一个参考系或坐标系中仍存在巨大挑战。
map-to-pan和构建基于图形而非线性参考基因组方法用于在泛基因组背景下总结归类SV信息。最近,一种基于线性和图形的参考基因组的混合方法开发出来。
转座子与作物改良的相关性
随着泛基因组在作物中的广泛应用,作为结构变异驱动因素的TEs在作物改良中将受到越来越多的关注。植物基因组(包括作物物种)中的TEs尤其普遍,TEs与作物表型的相关性已经被反复证明。转座因子可以以多种方式与功能相关,包括改变转录的基因产物的结构和数量(图2)。例如,在玉米中,一个Harbinger-like DNA转座子抑制ZmCCT9基因的表达,以促进长日照条件下的开花。在水稻中,Gypsy反转录转座子已被证明能增强OsFRDL4基因的表达并促进耐铝性。两个Copia反转录转座子独立插入到柑橘Ruby基因的启动子区域,导致其表达增强,并促进血橙性状的趋同进化。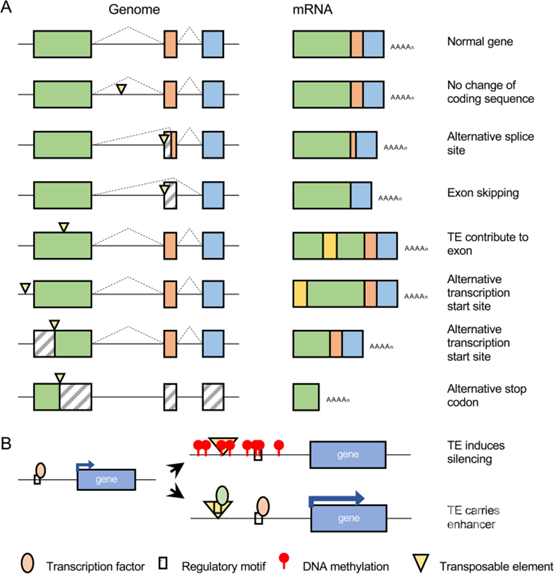
利用作物泛基因组进行QTL定位和GWAS研究
双亲群体的QTL定位和不同个体组成群体的GWAS定位被用来识别与期望表型相关的基因组区域。基于单个参考基因组的QTL定位或GWAS的一个主要缺陷是参考偏差。如果与一个性状相关的变异在参考基因组中不存在,那么QTL定位或GWAS将无法检测到它们(图3a,b)。例如,GWAS可以使用基于B73而非PH207基因组组装的标记来鉴定具有甘蔗花叶病毒抗性的玉米基因,因为该基因不存在于PH207组装中。随着种质资源更加多样化,这种局限性必将进一步扩大,使得很难确定的将有意基因资源纳入育种计划的种质资源中。另一个问题是,由于技术问题(例如,低序列覆盖率),相对于参考基因组的真实缺失与数据的缺失难以区分(图3c)。
迄今为止,QTL和GWAS的研究主要依赖于SNP数据,但其他标记在将不同类型的变异与表型联系起来方面也很有用。随着作物改良向全基因组角度的转变,SVs对性状变异的贡献越来越明显。最近在甘蓝型油菜中,利用从8个全基因组装配中鉴定出的PAVs进行了GWAS,发现了SVs与长角果长度、种子重量和开花时间之间的因果关系,而SNP-GWAS并没有捕捉到这些关系。同样,GWAS根据大豆泛基因组确定了与种子光泽变化相关的PAV。在桃中,也观察到了导致果实早熟、核仁周围果肉颜色、果实形状和扁平形状形成的候选SVs。然而,我们对SVs对表型性状变异的重要性的认识还处于起步阶段。预计在不久的将来会看到越来越多的SV潜在表型变异对作物改良的重要性。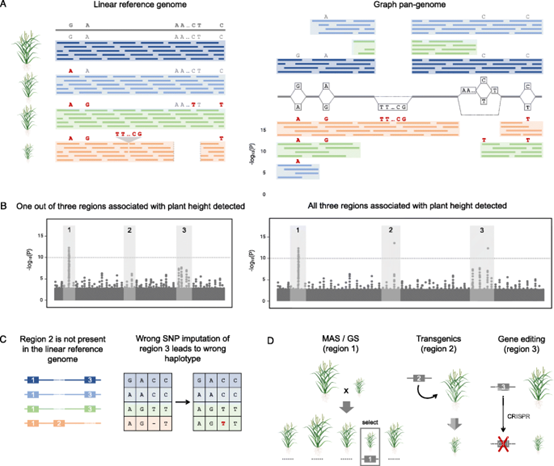 泛基因组学在作物改良中的挑战与机遇
泛基因组学在作物改良中的挑战与机遇
多倍体基因组的复杂性
异源多倍体和同源多倍体在植物中尤其常见。事实上,所有被子植物在进化史上至少经历了两轮多倍化事件。多倍化可以增加等位基因的多样性,扩大基因的互补性,产生新的表型变异,并有助于适应新的环境。利用这一点,植物育种家还产生了人工多倍体,从而提高了谷物产量、果实大小和无籽果实。
虽然多倍体作物对维持人类生命至关重要,但由于许多原因,对这些物种的基因组研究传统上非常具有挑战性。多倍体物种的高质量基因组组装较为困难。此外,多倍体作物的改良还面临着进一步的复杂问题:(1)当变异没有正确映射到亚基因组时,复杂性状的遗传结构的剖析可能会被混淆(2)生物学上,多倍体中更广泛的上位效应和亚基因组之间的调节反馈会使得基于基因型的表型预测复杂化。
测序技术和组装算法的进步已经解决了多倍体作物基因组组装的技术挑战。随着多倍体作物基因组研究的深入,由于基因组的冗余性和复杂性,多倍体作物内部的结构变异程度将大于二倍体作物,SVs可能是多倍体作物改良中特别富有成效的特征。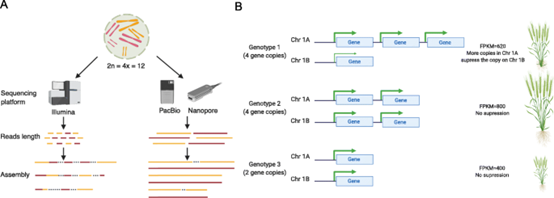
未研究作物的基因组资源
对于未研究的作物,由于这些物种的研究群体规模较小,并且在某些情况下,由于基因组复杂性带来的挑战,泛基因组辅助育种仍然有限。对于大多数未被研究的作物物种,转录组组装目前被用作基因组的替代物。虽然泛基因组研究在非模式作物中可能还处于起步阶段,但在模式作物中随着测序、组装算法和分析方法的快速发展以及成本的降低将很快使这项研究成为可能。从第一个水稻基因组组装的发表到第一个水稻泛基因组的发布,时间超过了十年。随着研究水平和技术的普及,包括泛基因组在内的基因组资源的开发必将更加迅速。
新物种和现存物种的快速驯化
高质量基因组和泛基因组的出现使作物驯化进入了一个新时代。利用泛基因组信息,育种人员可以更有效地识别驯化性状的遗传变异(如SNPs、CNV、PAV),并应用基因编辑工具在野生植物中快速获得理想的农艺性状。与野生近缘物种相比,驯化大大降低了作物的遗传多样性。鉴定和利用来自作物野生近缘种的遗传多样性一直是作物改良的一个主要思路。总之,泛基因组信息和CRISPR/Cas9技术能够实现野生植物的从头驯化,并可以减少使用来自第二和第三基因库(野生亲缘)的遗传变异障碍。
来源:frontiersin 植物科学最前沿
原文链接:http://mp.weixin.qq.com/s?__biz=MzIyOTY2NDYyNQ==&mid=2247507015&idx=5&sn=a730fe658c471a3d2352381817f89c9c
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn


