来源:植物科学最前沿
CRISPR/Cas9基因编辑系统中持续被关注的一个问题是gRNA的非特异性结合可能导致脱靶效应。然而,由于个体基因组的多样性,和短读长测序方法无法准确检测重复、低复杂度或AT/GC富集区域序列中的CRISPR/Cas9剪切效应,使准确预测CRISPR/Cas9的非靶活性具有一定的挑战性。
第三代测序是指单分子测序技术,具有读长长、单分子,无GC偏好性、测序过程无需进行PCR扩增、测序的同时可以检测碱基修饰的独特优势。目前,三代测序主要分为单分子荧光测序(SMS技术和SMRT技术)和纳米孔测序两种。其中,单分子实时测序技术(SMRT)基本原理是小孔捕获单分子DNA,并在DNA酶的作用下,使带有不同荧光基团标记的核苷酸结合到DNA序列上,通过统计荧光信号存在时间长短,区分匹配碱基与游离碱基,获得DNA序列。纳米孔测序技术(Nanopore sequencing)基本原理是使单个DNA分子的单个核苷酸逐一通过纳米孔,并且不同核苷酸通过时会有不同的电信号,通过检测电信号的差异,实现DNA测序。

近日,来自瑞典乌普萨拉大学Adam Ameur团队在Genome Biology在线发表题为“Amplification-free long-read sequencing reveals unforeseen CRISPR-Cas9 off-target activity”的文章。该文章提出了SMRT-OTS(基于SMRT技术的脱靶位点测序)和Nano-OTS(基于Nanopore sequencing技术的脱靶位点测序)两种新的、无扩增、长时间阅读的测序方案,用于体外检测CRISPR/Cas9系统的靶向和非靶向编辑效应(图1)。
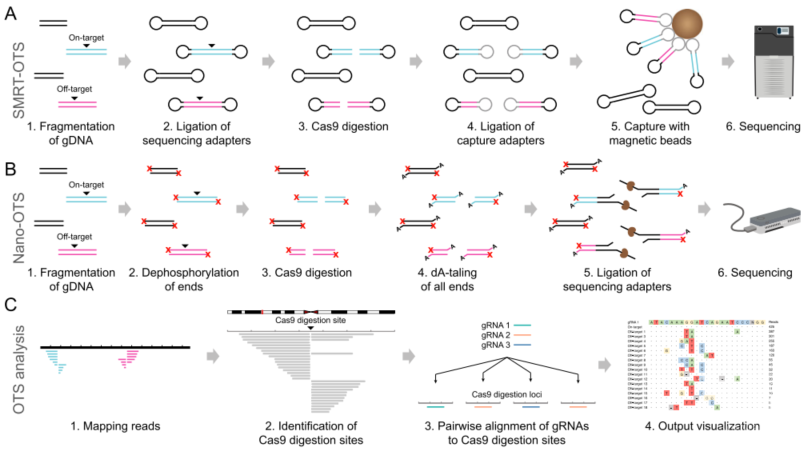
图1:图解SMRT-OTS和Nano-OTS检测Cas9切割位点方法
为了对上述两种脱靶位点测序技术进行评估,作者以人类细胞系HEK293为研究对象(图2),该细胞系已经具有18x覆盖度的高度精确的HiFi SMRT重测序数据。首先,利用三种不同gRNA靶向HEK293基因组DNA,分析发现两种方法共同检测到了55个高可信度的脱靶切割位点。其中,25个脱靶位点没有被脱靶预测软件所预测到,这是由于这些脱靶位点在参考的基因组中的序列与gRNA序列之间存在四个或更多的单核苷酸不匹配或插入/删除不匹配。另外的实验表明,85%的Cas9脱靶位点和在短读不能唯一匹配的基因体中可以检测到靶向点和非靶向点,是可以通过其他基于体外的检测方法找到。尽管SMRT-OTS和Nano-OTS在细胞中识别了几个先前验证过的脱靶编辑位点,但我们自己在人类成纤维细胞中的CRISPR/Cas9编辑实验中,并未在体外检测到预测位点产生可以被检测到的脱靶突变类型。然而,在靶向区域内存在大量的indel和结构变异位点。
总之,该文章提供了两种检测CRISPR/Cas9系统非靶向位点的方法,该方法具有无PCR扩增和读长长的特点。并且,可以检测到用脱靶预测软件无法预测的位点和实现在短读长测序无法准确测序的序列中的脱靶位点进行脱靶位点检测。
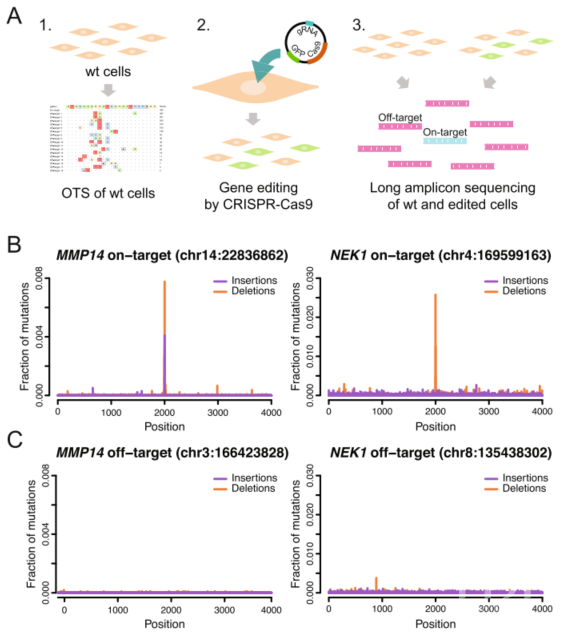
图2:CRISPR-Cas9编辑人类细胞的靶向和非靶向编辑位点分析
Tips: SMRT-OTS和Nano-OTS检测方法基本流程(图1)
SMRT-OTS方法是将随机剪断的DNA序列加上测序接头,之后用CRISPR/Cas9系统进行切割,并在切割位点处连接上捕获接头,之后磁珠可以捕获具有捕获接头的序列对其进行SMRT测序。
Nano-OTS方法将随机剪断的DNA序列的进行去磷酸化处理,去磷酸化后无法结合上接头序列,之后用CRISPR/Cas9系统进行切割,产生的断点序列再接上dA尾,使其可以接上测序接头对其进行纳米孔测序。
来源:frontiersin 植物科学最前沿
原文链接:http://mp.weixin.qq.com/s?__biz=MzIyOTY2NDYyNQ==&mid=2247504674&idx=6&sn=0c8c65e3218a4e6e5df34b3a813e5905
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn