常规核小体的结构包括一个由四种组蛋白H2A、H2B、H3、H4组装而成的蛋白核心,一条在组蛋白核心上缠绕1.6圈、长度为147 bp的双链DNA。核小体具有稳定的结构,对DNA组成和组蛋白修饰的改变均不敏感。组蛋白变体可改变核小体和染色质结构调控基因转录,在迄今测定的所有单核小体结构中,组蛋白H3变体核小体是构象改变最大的CENP-A核小体,结构显示,CENP-A核小体包含的DNA为121 bp,但是其蛋白核心结构变化不大。组蛋白H2A变体H2A.B和H2A.Z.2.2分别在精原细胞和人脑组织中特异表达,在精子发生、转录起始、RNA剪切等过程中发挥重要功能。H2A.B和H2A.Z.2.2形成开放的核小体结构,并“破坏”染色质结构,导致染色质松散。但是这种开放的核小体极不稳定,难以获得高精度结构。
近期,中国科学院生物物理研究所研究员周政课题组与研究员朱平课题组合作,在THE EMBO JOURNAL上,在线发表题为Structural basis of nucleosome dynamics modulation by histone variants H2A.B and H2A.Z.2.2的研究论文。该研究利用冷冻电镜,解析分别包含组蛋白变体H2A.B和H2A.Z.2.2的核小体结构,阐明H2A变体蛋白调控核小体动态性和染色质结构开放的分子机制。
为稳定H2A.B核小体,研究人员重组表达PARP1的DNA结合结构域肽段PARP1-DBD,并将其加入H2A.B核小体,用以制备冷冻电镜样品。生化及电镜结果显示,PARP1-DBD降低核小体DNA末端波动性,但是不影响核小体结构。在此基础上,研究人员解析分辨率为2.8 Å的H2A.B核小体结构。
与常规核小体相比,H2A.B核小体的蛋白核心发生明显构象变化,部分结构元件缺失。H2A.B-H2B二聚体间距增大,导致缠绕DNA螺距增大、核小体变得更厚。组蛋白核心上缠绕的DNA长度减少三分之一,仅为103 bp,导致其仅能缠绕蛋白核心1.2圈。此外,研究人员还解析H2A.Z.2.2核小体的3.9 Å电镜结构,结果显示,H2A.Z.2.2蛋白核心的构象变化与H2A.B核小体类似,但是该核心缠绕的DNA约为125 bp。Mnase酶切和八聚体组装结果表明,H2A.B与H2A.Z.2.2的C末端存在一个由6个氨基酸残基组成的八聚体组装调控序列(ROF,regulating-octamer-folding),H2A.Z.2.2独特的ROF序列可提高H2A.Z的酶促交换反应效率。该研究解析迄今发现的最为开放的核小体结构,揭示H2A.B和H2A.Z.2.2调控核小体结构变化与染色质动态性的分子机制。
朱平组助理研究员周旻和周政组博士后、特别研究助理戴霖昌为论文的共同第一作者,周政和朱平为论文的共同通讯作者。博士李成珉、博士研究生史刘歆和博士黄艳参与该研究。生物物理所生物成像中心为该研究提供冷冻电镜研究设备和技术支持,研究工作得到国家自然科学基金、科技部国家重点研发计划和中科院战略性先导科技专项(B类)等的资助。
论文链接
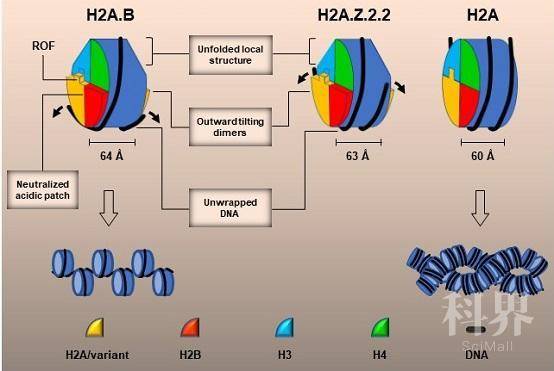
变体核小体结构与结构变化示意图
内容来源:中国科学院来源:中国科学院
原文链接:http://www.cas.cn/syky/202010/t20201022_4763913.shtml
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn




