来源:BioArt
核孔复合物(Nuclear pore complexes,NPCs)是核膜(nuclear envelope,NE)上的桥梁,负责核与胞质的物质交换。在动物体内,NPCs是由30个不同的核孔蛋白(nucleoporins,Nups)组装成的有110 KD的巨大装置。
其中,组装成内部富含FG-Nups(phenylalanine-glycinerich FG-Nups)的圆柱体的Nups被称为脚手架Nups(Scaffold Nups)【1-2】。FG-Nups在体外具有相分离的作用【3】,在体内也能使惰性分子不能渗透过圆柱体。
FG-Nups能和核转运受体(nuclear transport receptors,NTRs, 也称为输入蛋白importins, 输出蛋白exportins or 核转运蛋白karyopherins)瞬间作用形成复合物,并穿越NE。
转运方向受到小GTP酶 Ran的调控。原癌基因产物Ras类蛋白Ran(Ras-like nuclearprotein)是近期新发现的一种细胞周期和细胞核物质运输调控蛋白。RCC1,一种RanGTP交换因子(RanGTP exchange factor,RanGEF),可以维持核内高浓度的RanGTP。RanGTP置换掉输入复合物上的货物再一起随着输出复合物输出。
一旦到达胞质端,Ran GTP酶激活蛋白RanGAP(Ran GTPase activatingprotein)水解GTP并分解输出复合物【4-6】。RanGAP水解GTP需要结合Nup358。Nup358在低等生物不表达,但在动物体内发挥着重要的生物学过程,包括激活核转运,细胞周期运行,恶性肿瘤转移和病毒感染【7-10】。
NPC组装是个复杂的过程。在多细胞生物体内有两条经典的组装通路,分别发生在有丝分裂后期和分裂间期【11】。FG-Nups作为脚手架的中心,极易松散开,必须受到严格的调控以确保NPC组装的顺利进行【12】。
Non-NPC相关的Nups和importin β形成复合物,经RanGTP水解后释放Nups进行分裂间期和有丝分裂后的NPC组装【13-14】。在多细胞生物体,核孔也存在于内质网(endoplasmic reticulum,ER)的多层膜表面(stacked membrane sheets),称为环空板(annulate lamellae, AL),在配子和胚胎中尤为明显。以果蝇早期胚胎为例,AL插入核膜后能快速地为正在增殖的细胞核提供大量的膜和NPCs。AL也因此被认为是母系提供的NPC储存池【8】。但是,AL-NPC是如何能在非细胞核区域进行生物合成?又是谁在空间上调控这一过程?还不清楚。
2019年10月17日,来自德国欧洲分子生物学实验室的Martin Beck教授带领的团队在Cell发表题为“NuclearPores Assemble from Nucleoporin Condensates During Oogenesis”的长文,通过研究果蝇卵子发生的过程,发现了NPC在细胞核区域以外生物合成的新机制。这个过程需要Nups358等核孔蛋白凝结成不同的前体颗粒,沿着微管运动到AL处相互作用。此过程受到Ran和NTR的调控。

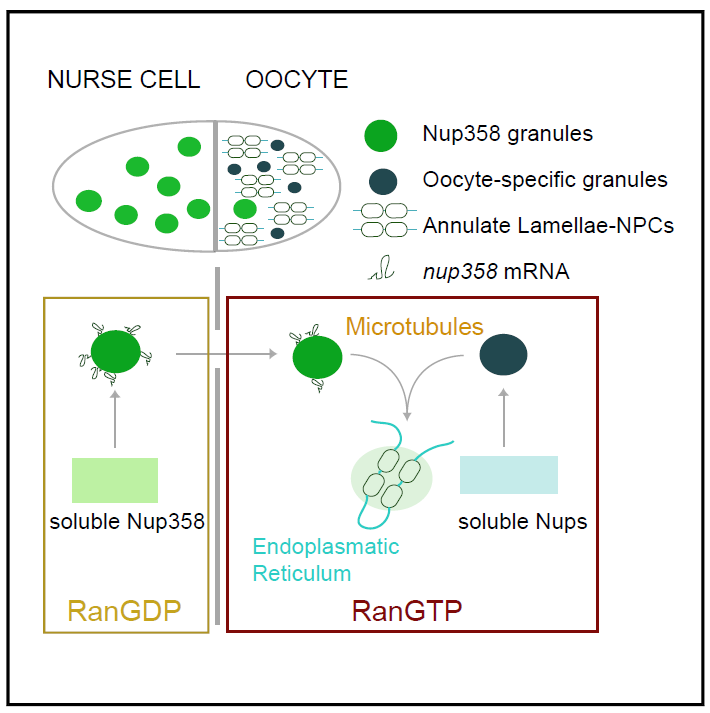
果蝇卵母细胞包含一种特殊的细胞,叫做抚育细胞(nurse cells), 果蝇卵母细胞的成熟过程是在细胞周期静止的情况下发生的,所以这是一个很好的研究非核区NPC组装的模型(图一)。作者们构建了特异性荧光标记脚手架Nups的RFP::Nup107果蝇,确定卵子发生过程中有AL合成。通过和表达GFP::Nup107雄果蝇交配,结果说明卵母细胞合成的AL为早期胚胎发育提供材料,而来自父系的AL在胚胎发育后期才合并入胚胎。说明AL合成是母系来源的。

图一
在卵母细胞AL-NPCs组装过程中,大多数Nup子复合物都会消失【8】,除了Nup358。说明Nup358在其中扮演了重要作用。作者们通过运用CRISPR/Cas9技术构建过表达GFP::Nup358果蝇来研究Nup358的定位,同时用FG-Nup marker 麦胚凝集素(Wheat Germ Agglutinin,WGA),发现Nup358与FG-Nup,AL,核膜共定位,说明其在NPC组装过程中的重要作用。
另外还发现,卵母细胞中除了有大小不一样的AL,还有两种不同大小的颗粒,一种是Nup358阳性的圆形颗粒,比AL还大还圆,并在局部呈现FG-Nup和脚手架共阳性。
Nup358在抚育细胞中非常普遍而在卵母细胞中则分布零星。另一种是小一点的Nup358阴性的颗粒,呈现FG-Nup阳性或脚手架阳性,称之为卵母细胞特异性颗粒(oocyte-specificgranules)。
对AL和两种颗粒的时空分布做了定量发现,AL会随着卵子发生不断积累,Nup358颗粒到卵子发生后期会消失,而卵母细胞特异性颗粒会一直存在到卵子发生的很后期。基于这两种颗粒的时间和空间分布,作者们猜测这两种颗粒可能是NPC生物合成的前体。
通过对活的卵母细胞进行跟踪和定量发现,Nup358颗粒从抚育细胞经过环形通道转移至卵质从而将抚育细胞的细胞质和卵母细胞进行连接;卵母细胞特异性颗粒要么直接和Nup358颗粒接触要么直接和AL接触,以保证材料运输的进行。两种颗粒之间的相互作用时间很短,呈一种动态。
卵母细胞活细胞实时成像结果显示,FG-Nups在AL上由于ER动态呈局部来回波动,而沿着微管运动时,他们表现出快速且有方向。如果通过喂食秋水仙素(colchicine)抑制微管的完整性,两种颗粒则不能进行融合,AL的合成也会减少。
以上实验结果说明,Nup358颗粒和卵母细胞特异性颗粒分别沿着微管运动到AL表面进行相互融合,作为后续的AL-NPCs生物合成的前体。
那么,这些颗粒是如何形成的呢?对Nup358颗粒的亚结构进行分析,发现里面聚集了许多其他类型的Nups,包括Nup214, FG-Nups和Nup gp210。已有文献报道多个Nups可以形成共凝结区域【15】。为了证实Nup358颗粒是否发生了凝结,作者们用凝结抑制剂1,6-环己烷处理卵母细胞,发现所有位置的颗粒的完整性都被破坏了。同时,后续的AL生物合成也被破坏。这些实验说明,Nups凝结成生物颗粒是AL生物合成所必需的。
Nups颗粒的不对称分布的原因是什么呢?果蝇卵母细胞具有极性,是由不同基因mRNA在不同位置富集程度决定的【16】。作者们利用单分子荧光原位杂交实验系统地研究编码Nup和NTR的mRNAs的定位。
结果发现,在检测的39个基因中中,nup358,nup153,ketel,karyopherin b3,和moleskin基因的mRNAs在AL,核膜和Nup358颗粒的表面都有富集。除了ketel,其他几个在抚育细胞中的表达比在卵母细胞中要多。
奇怪的是,在AL和Nup358颗粒相互作用区域,只有FG-Nups却没有nup358 mRNA。也就是说,nup358 mRNA在AL和Nup358颗粒相互作用区域转录成蛋白并进一步凝结,以供NPC生物合成所用。
为了证实这一点,作者们用翻译抑制剂环己酰亚胺(cycloheximide)处理卵母细胞后,nup358 mRNA则保留在AL和Nup358颗粒相互作用区域。这些实验结果说明,nup358mRNA以翻译依赖的方式定位到NPC 生物合成位点。
两条经典的NPC组装通路都由Ran调控【17】。作者们想知道卵母细胞中的AL-NPC生物合成是否也由Ran调控。和之前报道的一致,RanGAP在卵母细胞细胞核的表达和在抚育细胞核膜处差不多,但是RCC1在卵母细胞细胞核的表达则远远多于在抚育细胞的表达。
这也说明Ran在抚育细胞和卵母细胞的分布不一致从而在空间上指导AL生物合成。为了在体内进一步证实Ran调控AL生物合成。作者们分别在果蝇中过表达野生型的Ran,RanT24N突变体和RanQ69L突变体。RanT24N突变体能将Ran锁定在RanGDP状态而RanQ69L突变体能将Ran锁定在RanGTP状态。
结果和预想的一样,RanT24N突变体使Nup358凝结和AL生物合成都减少了。相反的,RanQ69L突变体使Nup358在抚育细胞就开始凝结,同时,AL也会在卵母细胞的极性聚集。如果用秋水仙素解聚微管,则AL的错误聚集会消失。这些实验都证实了,非核区的Nup358蛋白凝结和NPC生物合成也是由Ran调控。
总之,这篇文章首次发现了细胞核以外的NPC生物合成的具体机制。母系来源的AL在卵母细胞先合成,之后,RanGDP调控含有nup358 mRNA的Nup358颗粒沿着微管从抚育细胞转移到卵母细胞。Nup358颗粒和卵母细胞特异性颗粒在AL表面相互总用后,nup358等 mRNAs则转录成蛋白并凝结,从而进行NPC生物合成。
原文链接:
https://doi.org/10.1016/j.cell.2019.09.022
参考文献
1、Angelo, M.A., Anderson, D.J., Richard, E., andHetzer, M.W. (2006). Nuclear Pores Form de Novo from Both Sides of the NuclearEnvelope. Science 312, 440.
2、Beck, M., and Hurt, E. (2016). The nuclear porecomplex: understanding its function through structural insight. Nature ReviewsMolecular Cell Biology 18, 73.
3、Campbell, E.M., and Hope, T.J. (2015). HIV-1capsid: the multifaceted key player in HIV-1 infection. Nat Rev Microbiol 13, 471-483.
4、Dickmanns, A., Kehlenbach, R.H., and Fahrenkrog, B.(2015). Chapter Five - Nuclear Pore Complexes and Nucleocytoplasmic Transport:From Structure to Function to Disease. In International Review of Cell andMolecular Biology, K.W. Jeon, ed. (Academic Press), pp. 171-233.
5、Doucet, C.M., Talamas, J.A., and Hetzer, M.W.(2010). Cell cycle-dependent differences in nuclear pore complex assembly inmetazoa. Cell 141, 1030-1041.
6、Forler, D., Rabut, G., Ciccarelli, F.D., Herold,A., Köcher, T., Niggeweg, R., Bork, P., Ellenberg, J., and Izaurralde, E.(2004). RanBP2/Nup358 provides a major binding site for NXF1-p15 dimers at thenuclear pore complex and functions in nuclear mRNA export. Mol Cell Biol 24, 1155-1167.
7、Frey, S., Richter, R.P., and Görlich, D. (2006).FG-Rich Repeats of Nuclear Pore Proteins Form a Three-Dimensional Meshwork withHydrogel-Like Properties. Science 314,815.
8、Görlich, D., and Kutay, U. (1999). TransportBetween the Cell Nucleus and the Cytoplasm. Annual Review of Cell and DevelopmentalBiology 15, 607-660.
9、Hoelz, A., Debler, E.W., and Blobel, G. (2011). TheStructure of the Nuclear Pore Complex. Annual Review of Biochemistry 80, 613-643.
10、Knockenhauer, K.E., and Schwartz, T.U. (2016). TheNuclear Pore Complex as a Flexible and Dynamic Gate. Cell 164, 1162-1171.
11、Konishi, H., and Yoshimura, S. (2018). Non-FG-Nups,ELYS and Nup35, coordinate an ordered assembly of FG-Nups on the scaffold ofthe nuclear pore complex.
12、Matunis, M.J., Wu, J., and Blobel, G. (1998).SUMO-1 modification and its role in targeting the Ran GTPase-activatingprotein, RanGAP1, to the nuclear pore complex. J Cell Biol 140, 499-509.
13、Milles, S., Huy Bui, K., Koehler, C., Eltsov, M.,Beck, M., and Lemke, E.A. (2013). Facilitated aggregation of FG nucleoporinsunder molecular crowding conditions. EMBO Rep 14, 178-183.
14、Raices, M., and D'Angelo, M.A. (2012). Nuclear porecomplex composition: a new regulator of tissue-specific and developmentalfunctions. Nature Reviews Molecular Cell Biology 13, 687-699.
15、St Johnston, D. (2005). Moving messages: theintracellular localization of mRNAs. Nature Reviews Molecular Cell Biology 6, 363-375.
16、Ungricht, R., and Kutay, U. (2017). Mechanisms andfunctions of nuclear envelope remodelling. Nature Reviews Molecular CellBiology 18, 229.
17、Walther, T.C., Askjaer, P., Gentzel, M., Habermann,A., Griffiths, G., Wilm, M., Mattaj, I.W., and Hetzer, M. (2003). RanGTPmediates nuclear pore complex assembly. Nature 424, 689-694.
来源:BioGossip BioArt
原文链接:https://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652476753&idx=4&sn=b11ee9c1afcb0d1dfa58c4c03b5b53d0&chksm=84e206e5b3958ff3dacca48435ed6c31f752893c8fdbb6ddff9bbe3bfddda8967639fe77f44d&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn



