来源:宏基因组
哺乳动物的新陈代谢对内在和外在的变化都十分敏感,而生物钟和菌群是其中两项极为重要的因素,前者控制了食物何时摄取,而后者影响食物的消化吸收。具体来说,肠道菌群调控了宿主的代谢途径,并参与了发胖过程【1-4】,而宿主也会随着昼夜交替而规律性表达生物钟相关的分子【5-7】。作为宿主新陈代谢中最为基础的一环,何时进食与何时入睡相互影响,密不可分。那么,进食相关的肠道菌群与入睡相关的生物钟,这二者是如何相互影响,从而最终影响宿主新陈代谢呢?
来自美国西南医学中心的Lora V Hooper团队于2019年9月在Science上发表题为The intestinal microbiota programs diurnal rhythms in host metabolism through histone deacetylase 3 的文章,发现菌群可以诱导肠道上皮细胞表达组蛋白脱乙酰酶3(histone deacetylase 3,简称HDAC3),而HDAC3会按照昼夜节律被招募至染色体上,引导组蛋白依照昼夜节律发生乙酰化和去乙酰化,从而最终影响代谢相关基因的表达和食物的摄取。并且,HDAC3可以通过酯类转运分子Cd36促进脂类的吸收,从而导致肥胖。

多项报道指出,生物钟可以通过影响组蛋白的修饰分子,诱导代谢相关基因的表达【8-11】。所以,作者首先探讨,肠道菌群是否可以影响这些组蛋白修饰分子。白昼时间定为0点到12点,黑夜时间定义为13点到24点。作者首先采集了0点到24点的正常小鼠(conventional,简称CV)和无菌小鼠(Germ-free,简称GF)的肠道上皮细胞(intestinal epithelial cells,简称IELs),通过组蛋白表观组测序(组蛋白3的9位和27位赖氨酸乙酰化情况,histone H3 lysine 9/27 acetylation,简称H3K9ac和H3K27ac)两种检测方法,发现,CV小鼠的H3K9ac和H3K27ac呈现与生物钟相一致的节律变化,而GF小鼠的平均信号较CV小鼠强,但节律性却较弱。这些结果说明,肠道菌群可以调控上皮细胞以生物钟为规律的乙酰化。并且,还暗示,CV小鼠与GF小鼠的区别,很可能与脱乙酰酶(HDAC)相关。
接下来,作者试图寻找这一脱乙酰酶。作者首先检测了HDAC1-HDAC11这11种脱乙酰酶是否在肠道IELs中表达,并发现,HDAC1和HDAC3是表达量最高的两种。接下来,比较了CV小鼠和GF小鼠IELs中这两种脱乙酰酶的表达区别,发现,只有HDAC3在两种小鼠中表达量不同,GF小鼠中表达量较CV小鼠低。所以,HDAC3很可能就是作者想要寻找的脱乙酰酶。
随后作者进一步确定HDAC3的作用机制。因为在肝脏和脂肪中,HDAC3通常与其抑制因子(nuclear receptor co-repressor,简称NCoR)协同作用,行使功能【12-15】,所以,作者检测了IELs中二者的结合情况,发现在GF小鼠中,HDAC3与NCoR之间的结合要明显低于CV小鼠。接下来,作者构建了IELs特异性敲除HDAC3的小鼠,发现,这种小鼠的H3K9ac和H3K27ac与GF小鼠十分类似。上述结果说明,肠道菌群是通过HDAC3来调节组蛋白随着生物钟变化而变化的。
再下来,作者探讨了HDAC3对宿主新陈代谢的影响。作者发现,IELs特异性敲除HDAC3的小鼠血清中的酯类代谢产物浓度,不再随着生物钟而呈现节律变化。并且,生物钟相关的酯类代谢基因表达量降低,而参与酯类合成和吸收的基因表达量则上升,其中,就包括酯类转运分子Cd36。另外,作者通过给小鼠投喂抗生素清除其肠道菌群,野生型小鼠体重减轻,体脂含量也显著降低,但是其敲除HDAC3的同窝小鼠这两项指标则没有明显变化。这些结果说明,IELs的HDAC3可以调控酯类吸收,并且参与小鼠的发胖过程。
最后,作者探讨了HDAC3影响酯类吸收的机制。作者发现,上文提到的酯类转运分子Cd36在CV对照小鼠中的表达呈现节律变化,但是,在GF小鼠和IELs特异性敲除HDAC3的小鼠中,其表达量显著降低,并且,节律性变化也明显减弱模糊。这些结果说明,肠道菌群可以诱导Cd36的表达,并且,通过HDAC3影响酯类吸收。
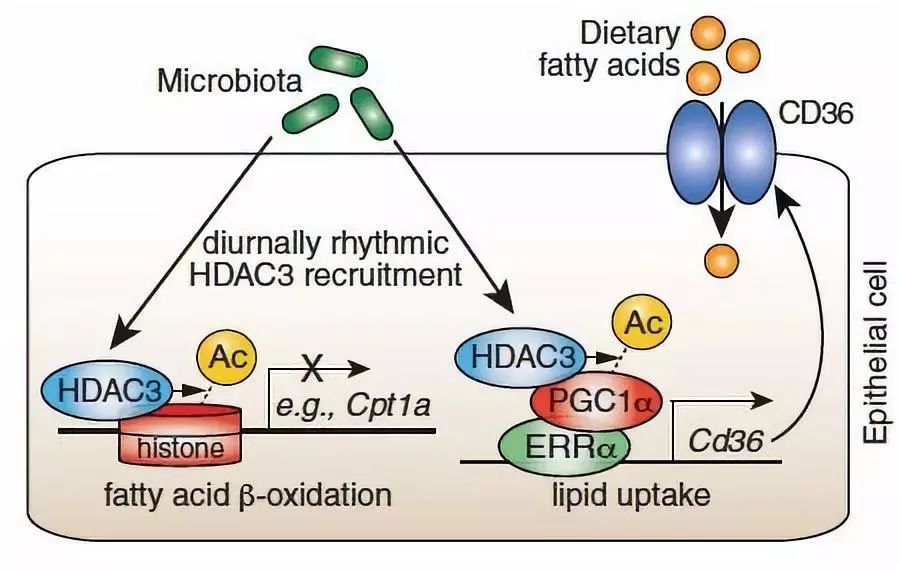
总的来说,肠道菌群和生物钟是目前生物学研究的两个热点,作者将二者联系起来,发现肠道菌群可以通过影响组蛋白去乙酰化酶,影响基因表达随着昼夜节律而变化,进而影响酯类吸收和宿主新陈代谢。
原文链接:
https://science.sciencemag.org/content/365/6460/1428
参考文献
1.P. J. Turnbaugh et al., Nature 457, 480–484 (2009).
2.F. Bäckhed et al., Proc. Natl. Acad. Sci. U.S.A. 101, 15718–15723(2004).
3.P. J. Turnbaugh et al., Nature 444, 1027–1031 (2006).
4.H. Tilg, A. Kaser, J. Clin. Invest. 121, 2126–2132 (2011).
5.C. A. Thaiss et al., Cell 167, 1495–1510.e12 (2016).
6.A. Mukherji, A. Kobiita, T. Ye, P. Chambon, Cell 153, 812–827 (2013).
7.Y. Wang et al., Science 357, 912–916 (2017).
8.M. Doi, J. Hirayama, P. Sassone-Corsi, Cell 125, 497–508 (2006).
9.A. Mehra, C. L. Baker, J. J. Loros, J. C. Dunlap, Trends Biochem. Sci. 34, 483–490 (2009).
10.D. Feng et al., Science 331, 1315–1319 (2011).
11.D. Feng, M. A. Lazar, Mol. Cell 47, 158–167 (2012).
12.Y. D. Wen et al., Proc. Natl. Acad. Sci. U.S.A. 97, 7202–7207 (2000).
13.L. Yin, M. A. Lazar, Mol. Endocrinol. 19, 1452–1459 (2005).
14.M. J. Emmett et al., Nature 546, 544–548 (2017).
15.M. J. Emmett, M. A. Lazar, Nat. Rev. Mol. Cell Biol. 20, 102–115 (2019).
来源:meta-genome 宏基因组
原文链接:http://mp.weixin.qq.com/s?__biz=MzUzMjA4Njc1MA==&mid=2247488907&idx=1&sn=9356c6970264b15b074024332cacecd1&chksm=fab9ff3acdce762c0a736c9e22aefe7cd393c1680bf273cbb7ff10ceb21329db593ea6de0850&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn


