来源:BioArt
100年前,研究人员发现染色体上有非常紧密的区域,并提出了异染色质结构这个概念【1,2】。100年后,研究人员进一步发现在多细胞生物染色体上,~25%-90%的染色体区域具有异染色质结构 【3,4】;并证明这些异染色质结构与基因组稳定、基因表达水平调控、细胞生长与分裂、细胞分化等直接相关 【5】。但是,经过100年的研究,产生组成性异染色质结构的根本机制还没有被确定。
DNA复制是核心生物事件。当DNA复制叉沿着染色体DNA移动并复制、合成DNA时,它会碰到许多内源的DNA复制叉停顿点。这些停顿点的大部分应该是由DNA二级结构导致的。各种DNA重复序列往往能形成多类型的DNA二级结构。人细胞染色体DNA大约有30-50万个复制叉停顿点。已证明停顿的DNA复制叉是不稳定的,需要严格的细胞调控,以维持其稳定、防止它垮塌,保持基因组完整性。也已证明Checkpoint(细胞周期检验点)是维持停顿复制叉稳定、防止其垮塌的一个必须细胞调控。如果停顿的DNA复制叉垮塌,将产生各种基因变异。大数据统计表明~66%的癌症是DNA复制错误产生的【6】。DNA复制叉垮塌被认为是造成DNA复制错误的主要原因。然而,在过去二三十年,该研究领域的进展不大,细胞调控维持停顿复制叉稳定的分子机制所知甚少。
2019年7月1日,PNAS在线发表北京大学孔道春团队题为Replication fork stalling elicits chromatin compaction for the stability of stalling replication forks的研究成果,揭示了异染色质结构形成与维持DNA停滞复制叉稳定息息相关。

研究发现,当DNA复制叉停顿后,停顿DNA复制叉周围的染色质结构变得更加紧密;并证明组蛋白H2BK33的去乙酰化、H3K9三甲基化等是复制叉停顿诱导的染色质结构变得更加紧密的一个重要方面。进一步实验确认组蛋白去乙酰化酶Clr6介导了复制叉停顿后组蛋白H2BK33的去乙酰化。Clr6通过Rad9Hus1-Rad1 complex (9-1-1 complex) 被招募到复制叉上。如果DNA复制叉停顿诱发的紧密染色质结构被破坏,DNA复制解旋酶将离开DNA复制叉,导致复制叉垮塌。研究还发现H2BK33的去乙酰化不受Checkpoint调控影响。
本工作发现了一种跟DNA复制检验点(checkpoint)平行的全新细胞调控机制。通过调控核小体,改变组蛋白修饰,在停顿的复制叉周围形成更紧密的染色体结构,从而防止复制叉垮塌。这一调控机制被命名为“The Chromsfork Control”:Chromatin Compaction Stabilizes Stalling Replication Forks (见下图) 。
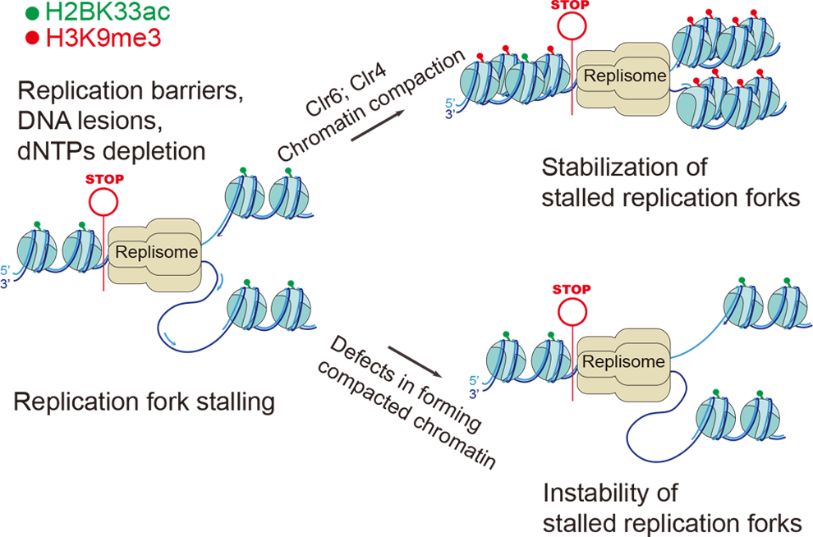
The Chromsfork Control调控模式图
孔道春实验室检查已知的异染色质区,几乎都存在DNA复制叉停顿/障碍点。他们进一步研究(未发表工作)也证明这些天然复制叉停顿点 (native replication barrier sites)能激活The Chromsfork Control,导致该区域染色质更紧密,变成异染色质区域。因此,基于这些发现,复制叉停顿诱发的染色质紧密结构应该是异染色质形成的最根本机制或根本机制之一。一旦异染色质结构的形成被起动,辅助一些其它生化机制,最终在某一个特定染色体区域形成异染色质结构。
The Chromsfork Control的发现,推动了对异染色质结构形成机制的理解,也将极大推动我们对细胞如何维持DNA复制叉稳定、保持基因组完整的分子机制理解。
据悉,北京大学孔道春教授为本文的通讯作者,北京大学博士后冯刚(现在福建医科大学独立研究基因组稳定性机制)和博士生袁越为本文的并列第一作者,北京大学博士生李泽阳、王露、张波、罗杰琛和北京大学纪建国教授对本文有重要贡献。
原文链接:
www.pnas.org/cgi/doi/10.1073/pnas.1821475116
参考文献
1. Montgomery TH. (1901), A study of chromosomes of the germ cells of metazoan. Trans Am Phil Soc. 20: 154-136;
2. Heitz E. (1928). Das heterochromatin der Moose. I. Jahrb Wiss Bot. 69: 762-818
3. Lander et al. (2001). Initial sequencing and analysis of the human genome. Nature, 409: 860-921
4. Vicient & Casacuberta (2017). Impact of transoposable elements on polyploidy plant genome. Ann. Bot. 120:195-207
5. Allshire & Madhani (2018). Ten principles of heterochromatin formation and function. Nature Reviews Molecular Cell Biology. 19: 229-244
6. Tomasetti et al. (2017), Stem cell divisions, somatic mutations, cancer etiology, and cancer prevention. Science, 355: 1220-1334
来源:BioGossip BioArt
原文链接:https://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652472226&idx=4&sn=372883c46093a9c27608273f2c9d5f0e&scene=0#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn



