内容来源:中国科学院
真核生物细胞器基因组包括线粒体和质体(包括叶绿体、白色体等)的全部DNA分子,是细胞质遗传的主要载体。在动植物和真菌的单个细胞内,有多个(甚至成千上万个)细胞器基因组单元的拷贝,使得利用低覆盖度的全基因组测序数据组装得到完整的细胞器基因组成为可能。随着DNA高通量测序技术的发展,测序成本下降,低覆盖度的全基因组测序数据得以大规模产生,如何快速、准确地组装细胞器基因组对后续生物学问题的研究非常重要。细胞器基因组序列在研究真核生物系统发育、谱系地理、杂交和物种鉴定等方面具有重要价值。自1986年Shinozaki等破解烟草叶绿体基因组以及近年来二代测序技术的发展,质体系统发育基因组已成为解析植物系统发育关系的手段之一。
中国科学院昆明植物研究所植物多样性与基因组学团队李德铢研究组和伊廷双研究组,关注基于植物质体基因组的系统发育、基因组结构演化和DNA条形码研究,发展基于质体基因组数据分析的研究体系,并取得重要进展(Ma et al., 2014. Systematic Biology;Zhang et al., 2017. New Phytologist;Li et al., 2019. Nature Plants;Zhang et al., 2020. Systematic Biology)。研究团队重视质体基因组分析方法的开发和应用,开发质体基因组注释软件PGA (Qu et al., 2019, Plant Methods)并得到广泛应用。
针对已有细胞器基因组组装软件组装流程的低效率、低成功率、低准确度,以及需要人工介入等问题,研究团队联合中科院西双版纳热带植物园和美国宾州州立大学,开发出一套细胞器基因组组装工具GetOrganelle,实现大规模细胞器基因组快速、准确地组装。GetOrganelle的核心流程包括:通过“种子”序列获得部分目标相关reads;延伸reads获得所有目标相关reads;对reads从头组装得到组装图形;过滤组装图形;识别细胞器组分并自动导出所有可能的细胞器基因组结构(图1)。GetOrganelle在baiting and iterative mapping的基础上提出reads预分群算法;提出适用于细胞器基因组的估算contigs拷贝数的算法,该算法能综合组装图信息和测序深度信息(图2)。基于50个植物物种的公开reads数据集的测试显示,在计算资源消耗略高的情况下,GetOrganelle默认参数的完整成环率(78%)高于工具NOVOPlasty最好参数的结果(16%);而资源消耗接近甚至更低的情况下,GetOrganelle仍能保持高于NOVOPlasty的成环率。并且,在K=23和K=31的情况下,NOVOPlasty约20%~25%的假阳性率(错误结果谎称完整成环)(图3)。在不同参数的测试下,GetOrganelle的结果一致性优于NOVOPlasty。Read mapping显示,GetOrganelle的结果准确性不仅高于NOVOPlasty,也高于基于相同reads数据的已发表结果(图4),并发现部分已发表结果的组装错误。在56个动物数据和50个真菌数据测试中,GetOrganelle获得比NOVOPlasty更高的线粒体基因召回率。此外,在Freudenthal et al. (2020) 针对主流叶绿体基因组组装工具(包括chloroExtractor,Fast-Plast,GetOrganelle,IOGA,NOVOPlasty,org.ASM等)的基准检测文章中,GetOrganelle获得高于其他工具的成环率和准确性,被推荐作为默认(组装工具)选项。
9月10日,相关研究成果以GetOrganelle: a fast and versatile toolkit for accurate de novo assembly of organelle genomes为题,在线发表在Genome Biology上。昆明植物所博士金建军和版纳植物园博士郁文彬为论文的并列第一作者,研究员李德铢和伊廷双为论文通讯作者。研究工作得到中科院战略性先导科技专项、国家自然科学基金重点国际(地区)合作研究项目、中科院大科学装置开放研究项目和中国西南野生生物种质资源库“交叉合作团队”开放研究项目的资助。
GetOrganelle的源代码于2016年4月在GitHub第一次上线。2018年5月,报道GetOrganelle的第一版预印稿在bioRxiv上线。2020年3月,GetOrganelle快装版在Bioconda上线。此外,GetOrganelle的动物meta-mitogenomics测试版已经上线,利用三代测序数据进行组装的功能正在开发中。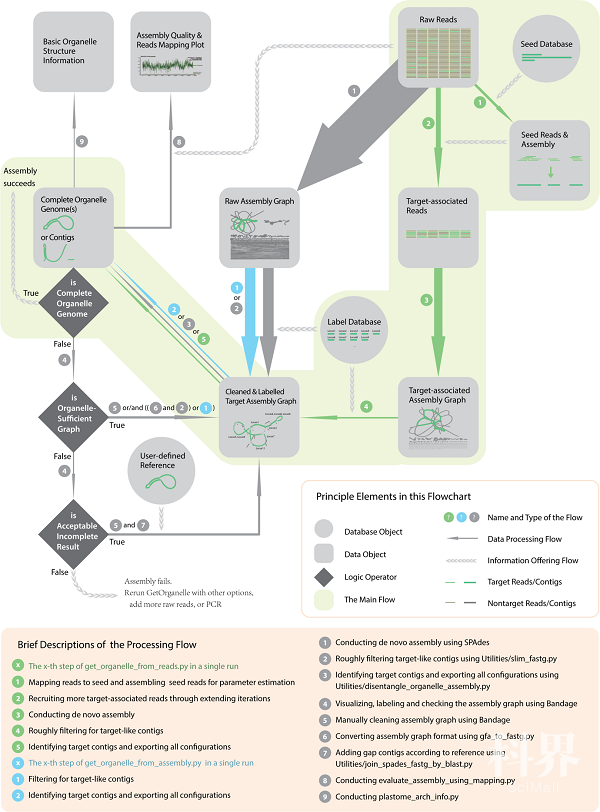
图1.GetOrganelle的工作流程图
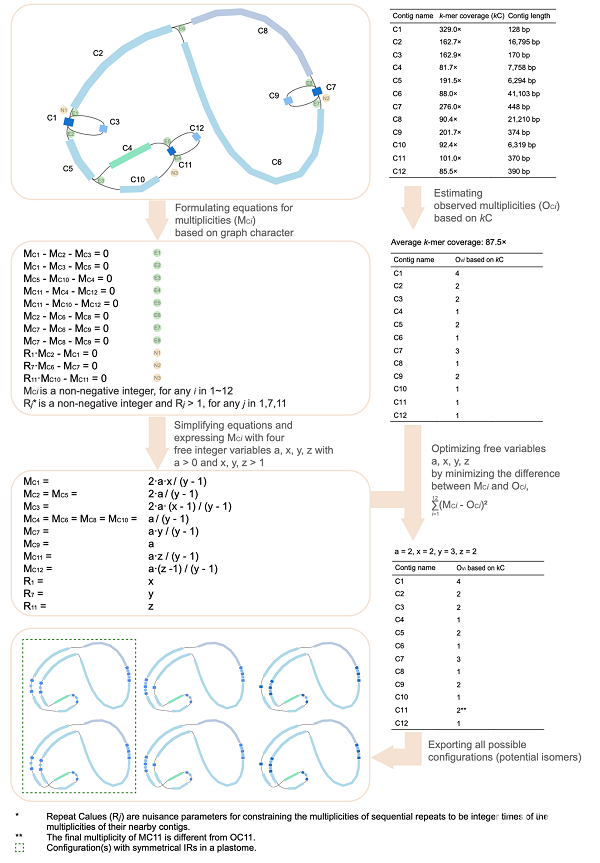
图2.GetOrganelle的contigs拷贝数估算及基因组结构导出算法示例
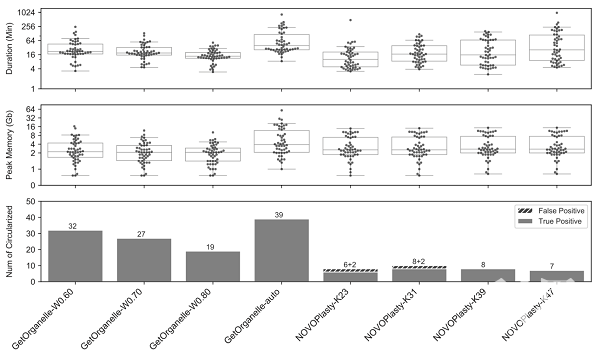
图3.GetOrganelle和NOVOPlasty分别在50个公开植物数据上的四组不同参数的测试结果
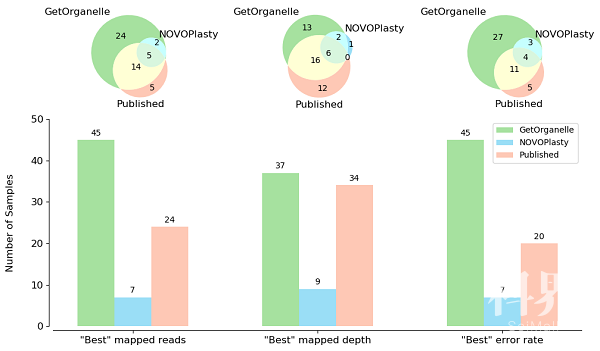
图4.基于Read mapping用50种植物的公开数据,评估并比较GetOrganelle组装质量、NOVOPlasty组装质量的和已发表的质体基因组的组装质量,统计三者在组装质量上最好(最多reads数、最高深度或者最低错误率)的样本个数。
来源:中国科学院
原文链接:http://www.cas.cn/syky/202009/t20200914_4759636.shtml
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn