来源:BioArt
在癌细胞中,一些抑癌基因的失活可能显著地改变细胞状态,导致癌细胞对某些药物异常敏感。例如,抑癌基因BRCA1/2失活后,PARP抑制剂可对癌细胞产生针对性的杀伤作用【1】,目前已在BRCA1/2突变的卵巢癌、乳腺癌和胰腺癌的临床治疗中取得了极好的效果。这种基于抑癌基因失活的“联合致死效应”是癌症治疗领域的一个重要研究方向。
2019年6月11日,中科院生化与细胞所姜海课题组在Cell Reports杂志上发表文章Systematic analysis of drug vulnerabilities conferred by tumor suppressor loss,系统性地分析了癌症中常见的近60个抑癌基因失活后,如何影响癌细胞对二十多种抗癌药物的敏感性,为基于抑癌基因失活发展治疗手段提供了新的研究方向。

在此前的研究中,分析“基因—药物”相互关系的一个重要方法是,测定近千种癌症细胞系的基因异变情况,并统计各个癌症细胞系对不同药物的敏感性,以此推断是否某个基因的异变会使细胞对某类药物异常敏感【2】。这一研究方法所生成的数据库GDSC(Genomics of Drug Sensitivity in Cancer; https://www.cancerrxgene.org/) 是目前癌症药物敏感性领域最大的数据库。然而,在抑癌基因—药物敏感性这一研究方向上,该方法显示了较大的局限性。在临床上已有确定结论的十余个抑癌基因—药物敏感性组合中,该方法仅能正确预测其中的两个。例如,该方法预测BRCA1/2突变后,癌细胞对PARP抑制剂产生抵抗性,这与临床结论相反。后续的分析发现,癌症细胞系的基因组和表达谱会发生较为迅速的改变,显著影响药物实验的准确性【3】,不同研究机构之间的数据可重复性较差【4】。此外,平均来说,两个癌症细胞系之间,会在数千个基因的突变、表达情况上存在显著差异。在这一背景下,将细胞系之间药物敏感性的差异归结于所聚焦的基因,存在一定的逻辑缺陷。
为了避免上述因素对于药物敏感性研究的影响,姜海课题组的此项研究中选用了一个基因组高度稳定的小鼠肿瘤细胞系,使用shRNA分别敲减了近60个重要的抑癌基因,并通过实验,一一分析了每一个抑癌基因的失活如何影响二十多种抗癌药物的敏感性。该实验系统所得出的数据具有高度的可重复性,所发现药物敏感性的变化也可以归结于目标基因的失活。这一研究发现,许多抑癌基因的失活都可能显著影响癌细胞对于一些药物的敏感性,这提示针对抑癌基因的失活,还有很多的探索针对性疗法的可能。此外,在该细胞系中所发现的抑癌基因—药物敏感性的变化在另一个肿瘤细胞系中的表现也高度一致,这提示,通过这一方法所发现的药物敏感性变化反映了基本的生物学机理。
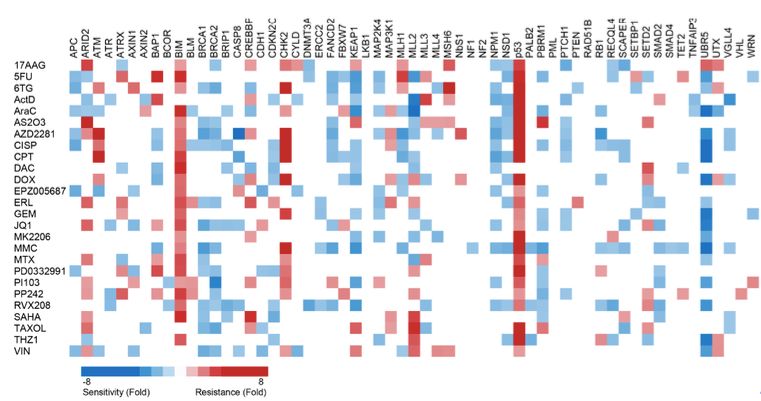
例如,该项研究发现,抑癌基因CREBBP的失活导致癌细胞对EGFR抑制剂产生抵抗。进一步的分析表明,CREBBP失活后,癌细胞发生EMT(上皮细胞—间充质转化),不再表达表皮细胞生长因子受体EGFR,因此EGFR抑制剂不再能够杀灭癌细胞。在靶向药物的临床治疗中,约20%左右的肿瘤最终发生EMT并耐药,然而分子机理尚不明确。该项研究为这一现象首次提供了机理明确的研究模型,并为EGFR抑制剂的耐药机理提供了新的探索方向。
此外,该项研究还发现,抑癌基因BAP1的失活导致癌细胞对DNMT1抑制剂高度敏感。在这一现象的提示下,研究发现BAP1是DNMT1的去泛素化酶,调控DNMT1的稳定性。在BAP1失活的肿瘤细胞中,DNMT1的蛋白水平显著降低,导致癌细胞对DNMT1的抑制剂更加敏感。这一发现也有助于进一步理解抑癌基因BAP1的功能以及DNMT1的调控机理。
综上所述,肿瘤中的基因变异如何影响药物敏感性是一个仍待解决的课题。由于实验系统的高度复杂性和不确定性,现有的基于上千个癌症细胞系的研究方法难以发现新的基因—药物相互作用关系。在本项研究中,初步记录了抑癌基因的失活如何影响药物的杀伤能力,在这些现象的基础上,我们可能更好地理解一些抑癌基因的生物学功能,并寻找针对抑癌基因失活的新的治疗手段。
原文链接:
https://doi.org/10.1016/j.celrep.2019.05.043
参考文献
1. Bryant, H.E., Schultz, N., Thomas, H.D., Parker, K.M., Flower, D., Lopez, E., Kyle, S., Meuth, M., Curtin, N.J., and Helleday, T. (2005). Specific killing of BRCA2-deficient tumours with inhibitors of poly(ADP-ribose) polymerase. Nature 434, 913–917.
2. Iorio, F., Knijnenburg, T.A., Vis, D.J., Bignell, G.R., Menden, M.P., Schubert, M., Aben, N., Gonçalves, E., Barthorpe, S., Lightfoot, H., et al. (2016). A Landscape of Pharmacogenomic Interactions in Cancer. Cell 166, 740–754.
3. Ben-David, U., Siranosian, B., Ha, G., Tang, H., et al. (2018). Genetic and transcriptional evolution alters cancer cell line drug response. Nature. 560(7718):325-330
4. Haibe-Kains, B., El-Hachem, N., Birkbak, N.J., Jin, A.C., Beck, A.H., Aerts, H.J.W.L., and Quackenbush, J. (2013). Inconsistency in large pharmacogenomic studies. Nature 504, 389–393.
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652471188&idx=7&sn=12dad49d6cd794be5cb5d5beb5588c1f&chksm=84e2ec20b3956536d4607831c1c889aa43d879073fd5d5ab69bf1e67f8040bb8ccfaf3ee5976&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn