来源:中国科学院昆明植物研究所
叶绿体是绿色植物细胞中进行光合作用的细胞器,是世界上成本最低、创造物质财富最多的生物工厂。叶绿体基因组主要编码与光合作用密切相关的蛋白质,作为细胞内半自主性的细胞器,叶绿体中也有许多蛋白质是核基因表达的产物,对叶绿体基因组的深入研究能够更好的理解核基因组与叶绿体基因组的遗传信息交流。叶绿体基因组是植物系统发育研究的重要数据来源,对于重建不同植物家系的演化历史提供了不可或缺的信息。高通量测序技术的快速发展使得叶绿体全基因组数据呈现爆炸式的增长,但其注释仍然是一个瓶颈,迫切需要一个用户友好型的工具对叶绿体基因组进行快速、准确、灵活的批量自动化注释。
中国科学院昆明植物研究所植物多样性与基因组学团队伊廷双和李德铢研究组多年来从事叶绿体系统发育基因组学研究。最近,研究组相关人员通过与美国Oberlin College的Michael J. Moore博士合作,开发了一个全新的叶绿体基因组注释软件PGA,即Plastid Genome Annotator(https://github.com/quxiaojian/PGA)。该软件克服了DOGMA、CPGAVAS、Plann、Verdant、GeSeq等现有叶绿体基因组注释软件存在的耗时耗力等问题,且能生成日志文档辅助用户检查注释结果。该软件具备三个新颖的特性。其一,采用了反向Blast搜索的方法确定基因在叶绿体基因组上的位置,不依赖庞大的数据库,显著提高了注释的速度;其二,软件包含新开发的基因和内含子特征边界检测算法,极大提高了基因和内含子边界注释的准确性;其三,作为一个本地注释叶绿体基因组的命令行工具,可以运行于任何配置Perl和Blast的计算机系统环境,同时可以自主选择叶绿体基因组参考序列,大大提高了叶绿体基因组注释的灵活性。
最近,该研究成果以“PGA: a software package for rapid, accurate, and flexible batch annotation of plastomes”为题在国际植物方法学重要期刊Plant Methods上在线发表。曲小健博士(现在山东师范大学生命科学学院工作)为论文第一作者,伊廷双研究员和李德铢研究员为共同通讯作者。该研究得到了中国科学院战略性先导科技专项(XDB31010000)、中国科学院西部之光西部青年学者项目(Y8247411W1)、国家自然科学基金国际(地区)重点合作研究项目(31720103903)和中国科学院重大科技基础设施开放研究项目(2017-LSF-GBOWS-02)的支持。
点击‘“阅读原文”查看文章链接。
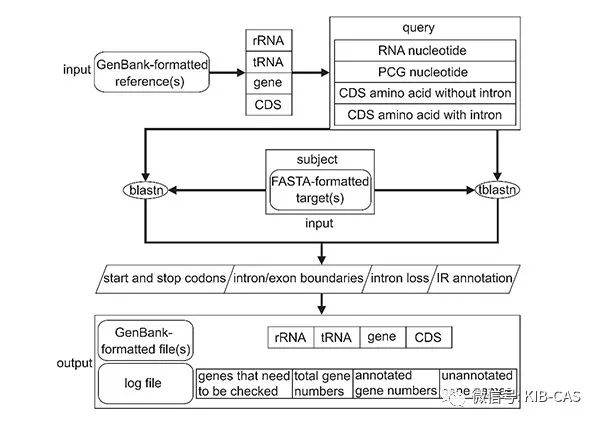
图1 PGA注释原理流程图
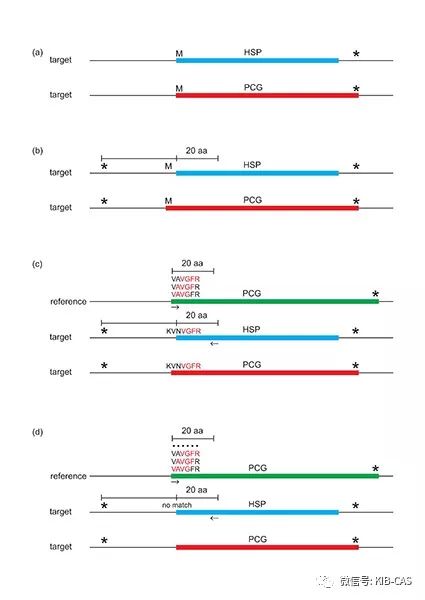
图2 PGA基因边界检测算法示意图
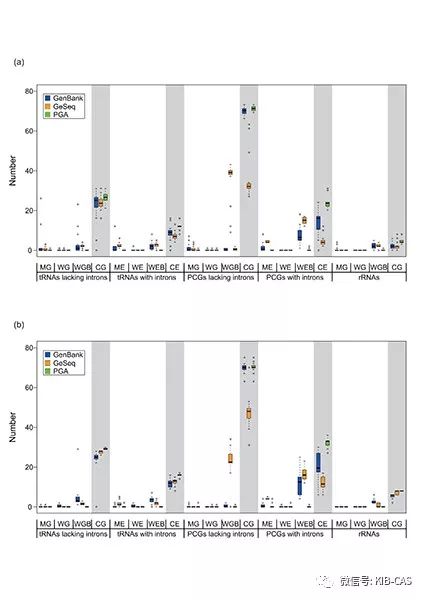
图3 PGA和GeSeq叶绿体基因组注释准确性比较
来源:KIB-CAS 中国科学院昆明植物研究所
原文链接:http://mp.weixin.qq.com/s?__biz=MzI2OTEwNDg0MA==&mid=2650200087&idx=1&sn=3bafdc6562f742e46674ee38ed07887b&chksm=f2e71bb0c59092a69cb4ce490ac6104d1c6defd66d7b73ee5d7287535fe471be87123ef90af8&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn


