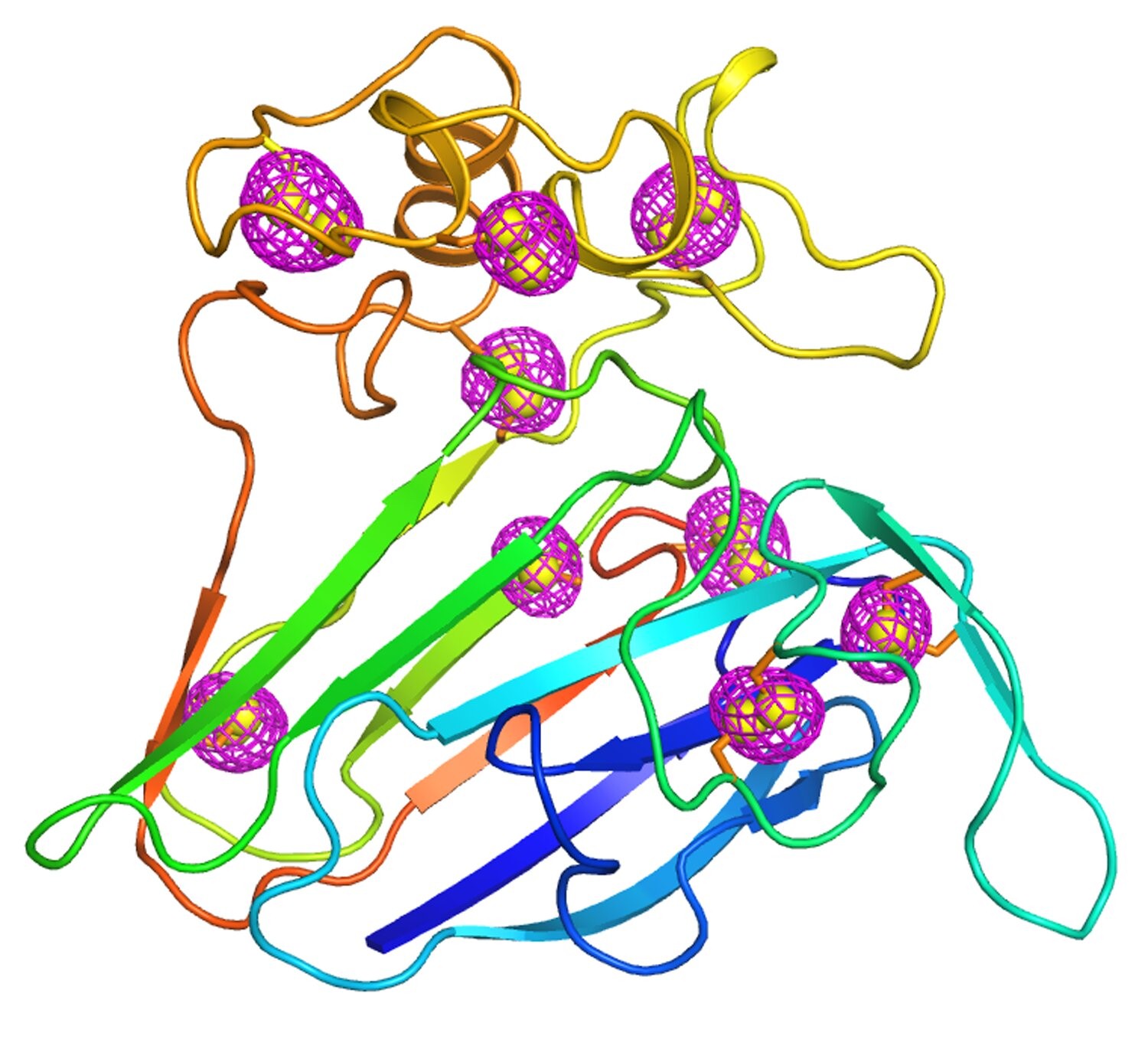
这幅图展示了一种经过深入研究的植物蛋白的结构,它是新开发的微晶体学技术的一个测试用例。紫红色的网状图案围绕着蛋白质固有的硫原子(黄色球体),显示了用低能量x射线衍射从数千个晶体中提取的异常信号。这些晶体不到十万分之一米,跟细菌差不多大小。
利用x射线揭示蛋白质的原子级三维结构,在了解这些分子如何在细菌、病毒、植物和人体中发挥作用方面取得了无数的进展,并指导了对抗癌症和艾滋病等疾病的精确药物的开发。但许多蛋白质无法在实验室条件下生长成足够大的晶体,这使其原子结构无法被破译。为了应对这一挑战,美国能源部布鲁克海文国家实验室的科学家和哥伦比亚大学的同事们开发了一种新的方法来解析微小晶体中的蛋白质结构。相关论文发表在5月3日的《国际联盟晶体学杂志》上。
这种方法依赖于独特的样本处理、信号提取和数据组装方法,以及能够将强x射线聚焦在布鲁克海文国家同步加速器光源II(NSLS-II)上,形成百万分之一米的光斑(大约是人类头发宽度的五分之一)的光缆。
“我们的技术真的打开了以前无法访问的大门,现在我们可以处理微晶核,包括难以结晶的细胞表面受体和其他膜蛋白、弹性蛋白以及许多复杂的人类蛋白质。” 这项研究的共同作者、布鲁克海文实验室科学家Qun Liu说,
自1958年以来,蛋白质晶体学一直是解决蛋白质结构问题的主要方法,随着时间的推移,x射线源变得越来越强大,蛋白质晶体学也得到了改进,使蛋白质结构的测定更加精确。为了确定蛋白质的结构,科学家们测量了类似于NSLS-II产生的x射线是如何衍射或反射到一个有序晶格中的原子上的,这个晶格由许多相同蛋白质拷贝以相同的方式排列而成。衍射图样传达了原子位置的信息。但这还远远不够。
Liu说:“探测器只记录衍射x射线‘波’的振幅,而不记录它们的相位(波与波之间的时间间隔)。”重建蛋白质的三维结构却两者都需要,这就是所谓的晶体相问题。”
晶体学家通过从另一种被称为反常散射的散射中收集相位数据,解决了这个问题。当比蛋白质的主要成分——碳、氢和氮,更重的原子吸收并重新发射某些x射线时,就会发生异常散射。当x射线能量接近那些重原子偏好吸收的能量时,就会发生这种情况。科学家有时会人为地将硒或铂等重原子插入蛋白质中。但自然出现在蛋白质分子中的硫原子,也能产生这样的信号,尽管强度较弱。
即使这些异常信号很弱,一个大晶体通常有足够的蛋白质拷贝和足够的硫原子使它们可以被测量。这为科学家们提供了精确定位硫原子位置所需的相位信息,并将衍射图转换成完整的三维结构。Liu说:“一旦知道了硫的位置,就可以计算其他蛋白质原子的相,因为硫和其他原子之间的关系是固定的。”
但是根据定义,微小的晶体没有那么多的蛋白质拷贝。因此,布鲁克海文/哥伦比亚大学的研究小组没有从单个大晶体中蛋白质的重复复制中寻找衍射和相位信息,而是开发了一种方法,可以从许多微小晶体中进行测量,然后收集集体数据。
为了处理这些微小的晶体,研究小组开发了带有微孔图案的样品网格。在将含有微晶的溶剂倒在这些安装好的网格上后,他们将溶剂除去,并将留在网格上的晶体冷冻起来。Liu说:“尽管如此,我们仍然面临着挑战,因为我们看不见网格上的微小晶体在哪里。”为了找到答案,我们利用NSLS-II的前沿微聚焦大分子晶体学(FMX)束线的微衍射技术对整个网格进行了测量。通过逐行扫描,我们可以发现这些晶体的隐藏位置。”
正如FMX束线的首席科学家Martin Fuchs所解释的那样,“FMX束线可以将x射线的全部强度聚焦到1微米(百万分之一米)大小。我们可以很好地控制光束的大小,使之与晶体的大小相匹配——目前实验中的晶体是5微米。这些能力对于获得最佳信号至关重要。FMX 束线的另一位科学家Wuxian Shi指出,“网格调查收集的数据包含了晶体位置的信息。此外,我们还可以看到每个晶体衍射的效果,这使得我们可以只选择最好的晶体来收集数据。”
然后,科学家们可以操纵样品夹,将每一个绘制出来的目的微晶片放回精确x射线束的中心进行数据收集。他们利用光束调谐时可用的最低能量尽可能接近硫原子的吸收能量,并收集异常散射数据。哥伦比亚大学的Wayne Hendrickson是这项研究的合著者之一。“幸运的是,NSLS-II是世界领先的同步加速器光源,能够提供明亮的x射线,覆盖广泛的x射线能量谱。尽管我们的能量水平略高于硫的理想吸收能量,但它产生了我们需要的异常信号。”
但是科学家们仍然需要做一些工作来提取这些重要的信号,并从许多微小的晶体中收集数据。“我们实际上得到了成千上万的数据,”Liu说。“我们使用了大约1400个微晶体,每个都有自己的数据集。我们必须把这些微晶体的所有数据放在一起。还必须剔除被强x射线破坏、或原子排列有微小变化的晶体。
“在被x射线破坏之前,单个微晶体对x射线衍射不足,不足以形成结构溶液,” NSLS-II的光子部副主任兼结构生物学项目经理Sean McSweeney说。“对于只有几微米大小的晶体,情况尤其如此。我们需要一种方法来解释这一点。”
他们通过复杂的多步骤工作流程来对数据进行筛选,丢弃可能由辐射损伤或不相容晶体引起的异常值,并最终提取异常散射信号。“这是关键的一步,”Liu说。“我们开发了一种计算程序,以确保只有兼容的数据以某种方式合并在一起,以使单个微晶体与衍射图形对齐。这为我们提供了确定结构所需的信噪比。”
这种技术可以用来确定任何难以结晶成大尺寸蛋白质的结构,包括使动物和植物等高级生命细胞表面受体。 Liu说:“为了通过进化适应环境,这些蛋白质是可塑的,有很多不均匀的修饰。在晶体中很难得到大量的重复拷贝,因为它们包装的不好。”
在人类中,受体是药物的共同目标,因此了解它们不同的结构有助于指导开发新的、更有针对性的药物。但这项技术并不仅限于小晶体。“我们开发的方法可以处理小的蛋白质晶体,也可以用于任何大小的蛋白质晶体,在任何需要结合来自多个样本的数据时都可以使用它,”Liu说。
科界原创
编译:花花
责编:张梦
期刊来源:《国际联盟晶体学杂志》
期刊编号:2052-2525
原文链接:
https://m.phys.org/news/2019-05-life-biochemical-networks-spontaneously-earth.html
版权声明:本文由科界平台原创编译,中文内容仅供参考,一切内容以英文原版为准。转载请注明来源科技工作者之家—科界App。






