来源:BioArt
撰文:Katherina
原标题:Nature亮点 | 全基因组CRISPR-Cas9筛选确认癌症治疗优先考虑的靶点
临床上,癌症病人体内肿瘤分子的特性决定了能否采用靶向治疗方案,从而提高治疗效果和降低毒副作用。然而,由于对潜在的治疗靶点认识不足,很多病人都无法使用靶向治疗的方案【1】。抗癌药物的研发也因为对这些靶点的性能和分子特性认识不足而阻力重重 【2】。
CRISPR-Cas9 基因编辑技术在生命科学领域已经得到了广泛的应用。单引导RNA(sgRNA, single-guide RNA)是由一小段自主设计的crRNA序列与tracrRNA序列支架融合得到的一个单个RNA分子。基于sgRNA库的CRISPR-Cas9筛选系统可以帮助我们了解到基因的功能和基因表达对细胞的作用 【3】。
2019年4月10日,来自英国维康桑格研究所(Wellcome Sanger Institute)的Mathew J. Garnett团队与Kosuke Yusa团队合作合作在Nature发表了题为Priorization of cancer therapeutic targets using CRISPR-Cas9 screens的论文,他们通过对324种肿瘤细胞系进行基因组范围的CRISPR-Cas9细胞功能筛选,整合分析,可以优先锁定潜在的癌症治疗靶点。
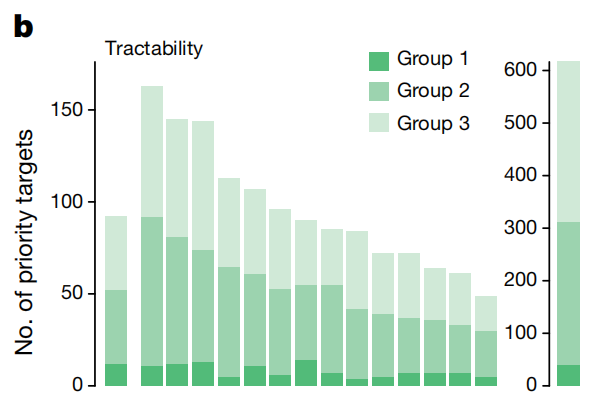
在此项研究中,研究人员首先通过CRISPR-Cas9的方法对339种肿瘤细胞系的18009个基因进行筛选,在满足条件的324种肿瘤细胞系中分析出哪些是对肿瘤细胞生长与存活重要的基因。而为了辨别这些基因哪些是特定环境下的功能基因(context-specific fitness genes),哪些是发挥核心作用的基因(core fitness genes),研究人员通过建立统计模型ADaM确定独立细胞系所需要的最少核心作用基因的数目。由此确定的肿瘤核心作用基因(pan-cancer core fitness genes)与之前提到的核心作用基因(core fitness genes)相比,哪个对重要的细胞进程需要承载发挥更多的基因功能。举例来说,在血癌细胞系中核心作用基因发挥着非常显著的作用。特定癌症型的基因相对应的在健康组织中也有极高的表达水平,我们若将其选为癌症治疗靶点,就会对健康组织细胞产生潜在的毒副作用。反之,在单一癌症型中有五个特定的核心作用基因,而这五个基因在对应的健康组织中表达水平很低或不表达,便意味着在组织细胞中这五个基因会诱发特定的癌症型。
随后研究人员建立了一种特定的计算模型框架,将特定环境下的功能基因通过CRISPR-Cas9的方法或基因生物标记筛选【4】,按照是否优先被考虑为癌症治疗靶点对肿瘤细胞系或特定癌症型的基因进行了从0到100的评分排序,其中排除了那些会造成潜在毒副作用的基因(评分为”0“)。进而,研究人员对这些可能作为癌症治疗的靶点,进行了药物制备可行性的评估,将这些靶点分为三组,优先考虑组1主要为丰富的蛋白质激酶作为药物研发的靶点。

许多癌症型都存在着肿瘤微卫星不稳定性,这是一种获得性基因特征暗示DNA修复机制的缺陷【5】。最终研究人员通过这种优先确认药物靶点的方法,锁定了维尔纳综合征 ATP依赖性解旋酶(WRN)是针对肿瘤微卫星不稳定性的重要治疗靶点,为这种癌症治疗靶点的筛选方法的应用和推广奠定了基础。
这项研究成果生动地展示出CRISPR-Cas9筛选癌症基因作为药物靶点并通过计算基因组学的方法辅助确认哪些基因应当优先作为抗肿瘤药物靶点,从而推动了药物研发初期阶段的进程。对于开发多样化、更加有效的新抗癌药物具有重要的现实意义。
原文链接:
https://www.nature.com/articles/s41586-019-1103-9
参考文献
1. Zehir, A. et al. Mutational landscape of metastatic cancer revealed from prospective clinical sequencing of 10,000 patients. Nat. Med. 23, 703–713 (2017).
2. Hay, M., Thomas, D. W., Craighead, J. L., Economides, C. & Rosenthal, J. Clinical development success rates for investigational drugs. Nat. Biotechnol. 32, 40–51 (2014).
3. Meyers, R. M. et al. Computational correction of copy number effect improves specificity of CRISPR–Cas9 essentiality screens in cancer cells. Nat. Genet. 49, 1779–1784 (2017).
4. Iorio, F. et al. A landscape of pharmacogenomic interactions in cancer. Cell 166, 740–754 (2016).
5. Ehrlich M, ed. DNA alterations in cancer: genetic and epigenetic changes. Natick, MA: Eaton Publ. p. 178. ISBN 978-1-881299-19-6. (2000)
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652468512&idx=1&sn=dffa8d7d848e3d2e53d83a3db8edd328&chksm=84e2e694b3956f82246d0cb16e203e7ddde2eacc83781665fba000e720b82473520ddeefca45&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn


