来源:核酸检测科学前沿

近日,来自英国Norwich研究院,在预印本杂志BioRxiv发表题为:“CoronaHiT: large scale multiplexing of SARS-CoV-2 genomes using Nanopore sequencing”文章,本文介绍了一种规模化SARS-CoV-2的纳米孔测序新研究,名为:CoronaHiT。
本文亮点:基于之前的报道,SARS-CoV-2的纳米孔测序(JoshQuick 2020)通量只有24个样本,作者利用一种新的转座酶引入接头和基于PCR加条形码技术实现了能够在单张纳米孔芯片上同时对48或94个SARS-CoV-2基因组进行多重测序,同时,作者还对比了CoronaHiT-48/94纳米孔测序技术与Illumina和ARTIC-ONT测序技术的差异比较。
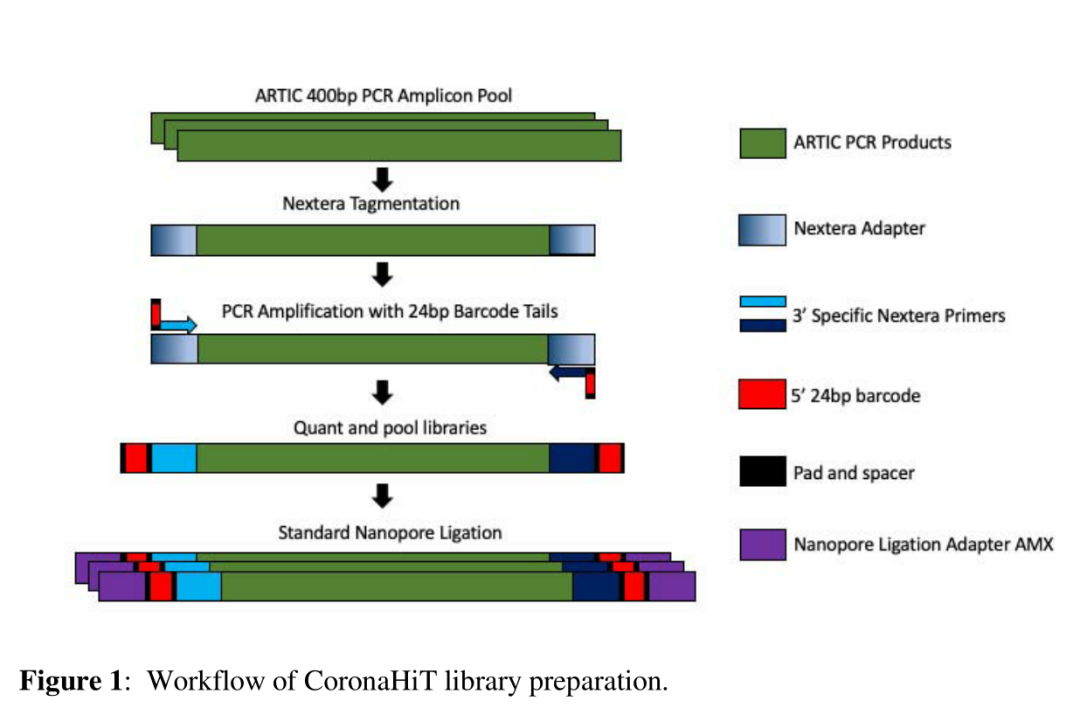
CoronaHiT的一个重要特点是不需要复杂的文库制备。该方法只需要10分钟的标记和30分钟的PCR,在两个步骤中一次对94个文库进行barcode标记,该过程不超过45min。
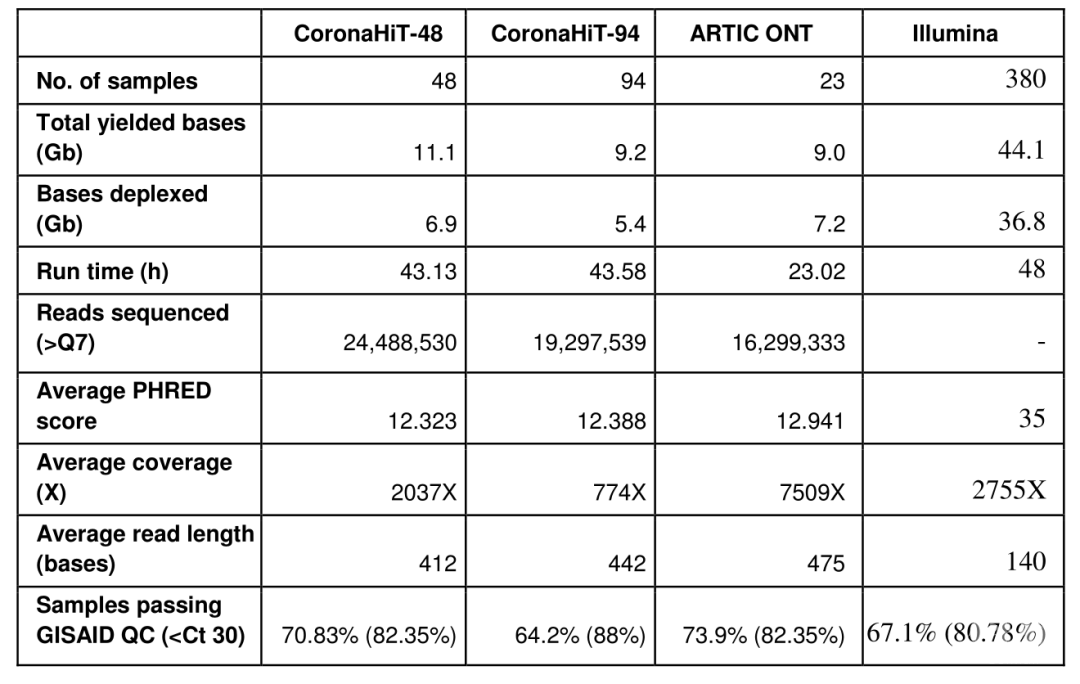
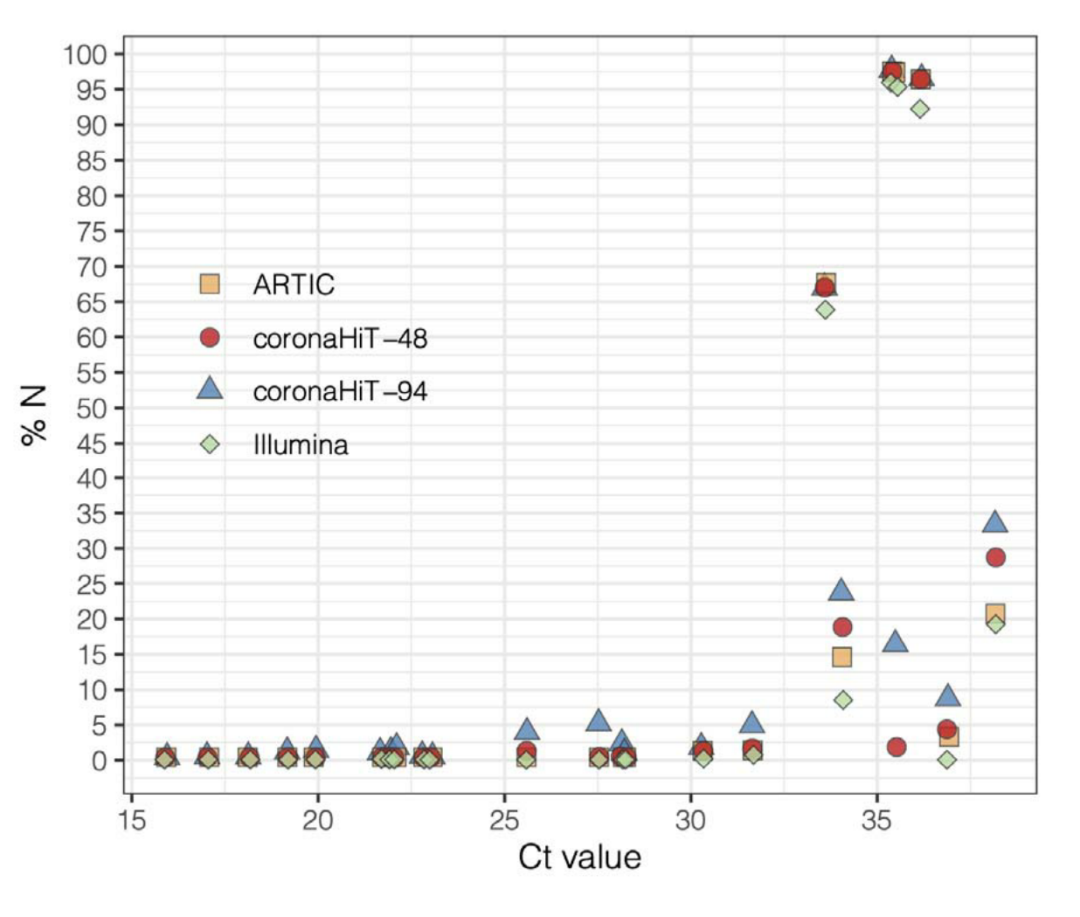
Coonahit-48、CoronaHiT-94与Illumina和ARTIC-ONT测序相比。当SARS-CoV-2阳性临床标本RNA提取液的循环阈值(Ct)>=32时,所有测序方法都会产生不准确的基因组。由23个具有广泛Cts范围的样本组成的同一组样本的结果表明,Coonahit-48、CoronaHiT-94、Illumina和ARTIC-ONT的一致基因组覆盖率(GISAID标准)分别为78.2%、73.9%、78.2%和73.9%,并且在最大似然树上具有相同的聚类。
总之,CoronaHiT可以在不影响所得到的基因组质量的情况下,对每纳米孔芯片能够提供高达94个SARS-CoV-2基因组的大规模测序,该技术有望推广到全球范围内的SARS-CoV-2基因组测序。
来源:hsjsqy 核酸检测科学前沿
原文链接:http://mp.weixin.qq.com/s?__biz=MzA5NTY3NDQ2MQ==&mid=2247485518&idx=1&sn=ffdb3b850d3a27f1329a44e4f3effe8a&chksm=90ba817ca7cd086ae6863b5e27c17a6ee085c265ae6fe91bb0db447364b1619002809c516de9&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn



