来源:BioArt
撰文 | 十一月责编 | 兮
密码子最优性(Codon optimality)是mRNA降解的主要决定因素,非最优密码子在转录本中的比例决定了mRNA半衰期的长短【1】。半衰期较短的、富含非最优密码子的mRNA的降解需要Ccr4-Not(Carbon catabolite repressor 4-negative on TATA)复合物的参与。Ccr4-Not复合物已知的主要功能与mRNA降解过程相关,该复合物作为主要的胞质3’-poly(A)尾巴的脱腺苷酶起始大多数mRNA的降解。但是关于Ccr4-Not复合物是如何协调密码子最优性监督过程以及mRNA降解过程具体分子机制的研究仍不清楚。
为了揭开Ccr4-Not复合物在监督密码子最优性过程中的作用,近日,德国慕尼黑大学Roland Beckmann、日本东北大学Toshifumi Inada以及美国凯斯西储大学Jeff Coller三个研究组在Science合作发文题为The Ccr4-Not complex monitors the translating ribosome for codon optimality,从冷冻电镜超分辨率结构出发,对翻译过程中核糖体对最优性密码子检测的具体分子机制进行了解析。
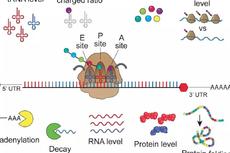
由于非最优密码子影响解码动力学并且mRNA发生共翻译降解【2】,因此可以推断密码子的最优性监测可能是直接发生在核糖体上的。核糖体中包含3个RNA结合位点,分别是A位点(Aminoacyl site)、P位点(Peptidyl site)和E位点(Exit site):A位点是结合氨酰基-tRNA的部位;P位点结合肽基-tRNA;E位点结合游离的tRNA,使核糖体变构,A位点打开。Ccr4-Not复合物与核糖体之间的相互作用以前曾经被报道过,该复合物中的Not4亚基作为E3连接酶能够对40S核糖体亚基蛋白进行泛素化修饰,而Not5亚基对于酵母中mRNA的可翻译性和多核糖体水平具有非常关键的作用【3,4】。
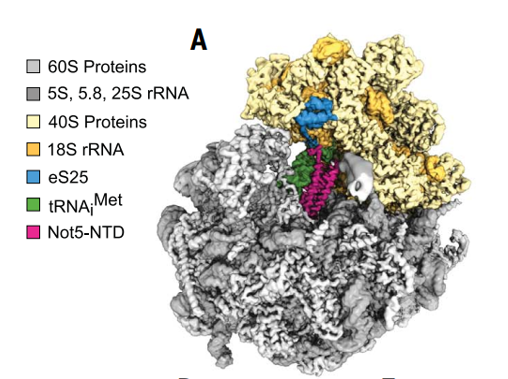
为了揭开核糖体与Ccr4-Not复合物之间的直接作用关系,作者们以标记的Not4为诱饵从酵母中共纯化了与核糖体结合着的内源的Ccr4-Not复合物。通过单颗粒冷冻电镜对单体部分进行分析后,作者们得到了一个总分辨率2.8 Å的结构图,包括A位点空置的核糖体80S亚基;位于P位点的tRNA;以及E位点存在的α-螺旋(图1)。该高分辨率的结构解析图使得作者们能够对Ccr4-Not复合物和核糖体之间的相互作用进行进一步地分析。Not5是Ccr4-Not复合物进化上保守的一个组分,在控制mRNA稳定性中具有重要作用【5,6】。作者们发现是Not5将Ccr4-Not复合物锚定到核糖体上的,并且Not5中包含3个α-螺旋的NTD(N-terminal domain)结构域对于Ccr4-Not复合物与核糖体的相互作用非常关键,Not5 NTD结构域的缺失会导致Ccr4-Not复合物与核糖体稳定相互作用的丢失。
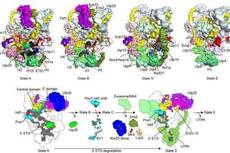
进一步地,作者们通过实验发现Ccr4-Not复合物同时与启始和延伸核糖体相互作用。在这两种情况下,Not5只有在核糖体A位点缺乏适合tRNA适合构象也就是解码动力学出现问题的时候才会到E位点之上(图2)。核糖体分析(Ribosome profiling)发现低最佳性的密码子会富集到被Ccr4-Not复合物结合的延伸核糖体中的A位点上。这也解释了冷冻电镜中tRNA在A位点占据的可能性较低的原因,同时也说明了密码子最优性的监控机制。在敲除Not5之后会造成mRNA降解复合物对感应密码子最优性的感知能力的丧失。
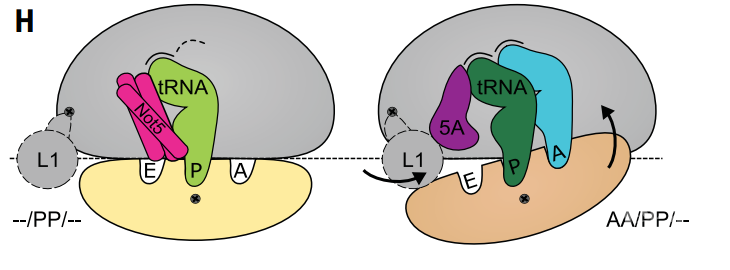 图2 eIF5A与Not5之间亚基转动以及竞争的示意图
图2 eIF5A与Not5之间亚基转动以及竞争的示意图总的来说,作者们的工作揭示出当非最佳密码子出现由于A位点缺乏tRNA导致低解码动力学的情况时,Ccr4-Not复合物通过Not5亚基特异性地结合到核糖体E位点,调节mRNA的降解过程。核糖体的这种状态是在A位点存在非最优密码子的情况下发生的,这也就解释了富含非最优密码子转录本半衰期较短的原因。该高分辨率的结构生物学结果将为进一步认识通过Ccr4-Not复合物为翻译效率与mRNA稳定性的协调提供了具体的分子和物理结构方面的机制上的见解。
原文链接:https://science.sciencemag.org/content/368/6488/eaay6912
制版人:琪酱
参考文献
1.Presnyak, V. et al. Codon optimality is a major determinant of mRNA stability. Cell160, 1111-1124, doi:10.1016/j.cell.2015.02.029 (2015).2.Tesina, P. et al. Structure of the 80S ribosome-Xrn1 nuclease complex. Nature structural & molecular biology26, 275-280, doi:10.1038/s41594-019-0202-5 (2019).3.Preissler, S. et al. Not4-dependent translational repression is important for cellular protein homeostasis in yeast. The EMBO journal34, 1905-1924, doi:10.15252/embj.201490194 (2015).4.Panasenko, O. O. & Collart, M. A. Presence of Not5 and ubiquitinated Rps7A in polysome fractions depends upon the Not4 E3 ligase. Molecular microbiology83, 640-653, doi:10.1111/j.1365-2958.2011.07957.x (2012).5.Muhlrad, D. & Parker, R. The yeast EDC1 mRNA undergoes deadenylation-independent decapping stimulated by Not2p, Not4p, and Not5p. The EMBO journal24, 1033-1045, doi:10.1038/sj.emboj.7600560 (2005).6.Alhusaini, N. & Coller, J. The deadenylase components Not2p, Not3p, and Not5p promote mRNA decapping. RNA (New York, N.Y.)22, 709-721, doi:10.1261/rna.054742.115 (2016).
来源:BioGossip BioArt
原文链接:https://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652487596&idx=6&sn=f89e44f62ce95c621e0b074a23992149&chksm=84e22c18b395a50ea8f125d5c8ac3644b519998279f72bddaa1a1849665c67c279c36869e771#rd
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn