来源:BioArt
责编 | 兮
自CBE被报导以来,多种策略对其进行了优化改进。例如在提高活性方面,主要是通过密码子优化及增加更强的核定位信号【4】、用高活性的胞嘧啶脱氨酶APOBEC3A或 Anc689替换 APOBEC1【4,5】、融合额外的 UGI 增加编辑效率和产物纯度【6,7】。而通过对脱氨酶的分子进化获得的evoAPOBEC1-BE4max大幅提高针对GC motif中胞嘧啶的编辑效率【8】。虽然这些方法一定程度上提高了CBE的活性,但是对于编辑窗口的影响不大,特别是更靠近PAM序列的碱基仍然很难被编辑到。因此,是否有新的策略可以提高编辑活性而又能扩增靶向碱基的范围,一直是碱基编辑器优化的难点。
2020年5月11日,华东师范大学李大力课题组在Nature cell Biology发表了题为Increasing the efficiency and targeting range of cytidine base editors through fusion of a single-strand DNA binding protein domain的研究论文,报道了一系列超高活性的胞嘧啶碱基编辑器(hyCBE),证实其提高编辑效率、扩展编辑窗口的同时保持高效精准的工作性能。

由于CBE主要是以单链DNA(ssDNA)为底物,因此研究团队猜测如果增强脱氨酶与ssDNA的结合能力,是否能增加脱氨酶作用时间而增强活性呢?于是,通过对10个非序列特异性的ssDNA结合结构域(ssDBD)与CBE进行融合,通过筛选,发现将Rad51蛋白的ssDBD融合到APOBEC1与Cas9n之间能显著提高碱基编辑活性,同时编辑窗口也大幅增加(图1)。通过10个靶点的分析,在编辑窗口C4-C8中,hyBE4max比BE4max活性提高了1.5-2倍,而在C9-C15这些更靠近PAM的碱基中,活性最多提高了18倍。
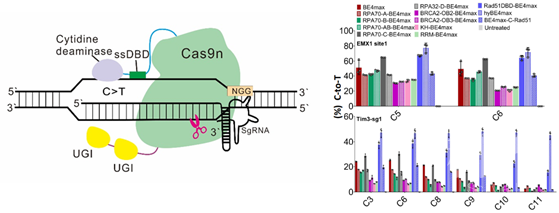 图1. hyCBE模式图及对不同ssDBD的筛选结果
图1. hyCBE模式图及对不同ssDBD的筛选结果为了验证融合ssDBD的策略是否具有通用性,用类似的方法改造A3A-BE4max和特异性识别TC motif中C碱基的eA3A-BE4max, 获得了hyA3A-BE4max 和 hyeA3A-BE4max。通过大量的实验证明,相比A3A-BE4max,hyA3A-BE4max活性在C3-C11位点提高了1.2-2倍,C12-C17位点活性提高3-4倍,通过小鼠胚胎显微注射也进一步证明其在编辑更靠近PAM序列的碱基的独特优势。与eA3A-BE4max 相比,hyeA3A-BE4max仍然能非常特异性地靶向TC motif中的C,编辑窗口由C4-9拓展为C4-15,活性最高提高了 257倍;而在小鼠胚胎中靶向Dmd基因在C13位编辑效率也大大提高,F0小鼠平均编辑活性提高了近60倍,F0代小鼠获得DMD纯合点突变的效率达到40%。
论文进一步验证hyCBEs特别是hyeA3A没有检测到DNA或者RNA层面的脱靶,具有非常高的精准性。最后,通过比较多种CBE在编辑胎儿血红蛋白(HBG1/2)启动子-117位点的能力发现,hyeA3A能在红细胞前体细胞系中精确催化-117G>A的转换,而周围同时发生碱基突变(bystander mutation)的细胞相比,具有更高的HBG表达水平,展示了 hyeA3A-BE4max 对于精准治疗β-血红蛋白病的巨大潜力。该工作开发了能提高编辑活性拓宽靶点范围的一系列新的CBE,为基础研究与基因治疗提供了新的优化工具。
华东师大生命科学学院2016级博士研究生张晓辉,2018级硕士研究生陈亮和2017级博士研究生朱碧云为该论文的共同第一作者,华东师范大学为第一作者单位,李大力教授为本文通讯作者,刘明耀教授对本课题给予了悉心的指导,同济大学毛志勇教授课题组在实验室提供了重要支持。
原文链接:https://doi.org/10.1038/s41556-020-0518-8
制版人:琪酱
向上滑动阅览参考文献
1.Rees, H.A. & Liu, D.R. Base editing: precision chemistry on the genome and transcriptome of living cells. Nat Rev Genet 19, 770-788 (2018).
2.Ran, F.A. et al. Genome engineering using the CRISPR-Cas9 system. Nat Protoc 8, 2281-2308 (2013).
3.Komor, A.C., Kim, Y.B., Packer, M.S., Zuris, J.A. & Liu, D.R. Programmable editing of a target base in genomic DNA without double-stranded DNA cleavage. Nature 533, 420-424 (2016).
4.Koblan, L.W. et al. Improving cytidine and adenine base editors by expression optimization and ancestral reconstruction. Nat Biotechnol 36, 843-846 (2018).
5.Wang, X. et al. Efficient base editing in methylated regions with a human APOBEC3A-Cas9 fusion. Nat Biotechnol 36, 946-949 (2018).
6.Komor, A.C. et al. Improved base excision repair inhibition and bacteriophage Mu Gam protein yields C:G-to-T:A base editors with higher efficiency and product purity. Sci Adv 3, eaao4774 (2017).
7.Wang, L. et al. Enhanced base editing by co-expression of free uracil DNA glycosylase inhibitor. Cell Res 27, 1289-1292 (2017).
8.Thuronyi, B.W. et al. Continuous evolution of base editors with expanded target compatibility and improved activity. Nat Biotechnol 37, 1070-1079 (2019).
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652484455&idx=1&sn=1989ea6d4453fdaa583eb352c421d5d2&chksm=84e220d3b395a9c54d7b6ffc22d479e5aa6e470885456d16feef72cdfa7f42d5ad60b5aea618&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

