来源:BioArt
责编丨兮目前,由新型冠状病毒SARS-CoV-2引起的COVID-19迅速发展成为全球性流行性疾病。尽管疫情暴发至今已有2月余,市面上仍然缺乏用于特异性预防和治疗COVID-19的药物。阻断性单克隆抗体具有极高的抗原特异性,是中和病毒感染的首选方案之一【1,2】。因此,制备能够特异性阻断SARS-CoV-2病毒进入宿主细胞的单克隆抗体,可高效地预防和治疗COVID-19。最新研究表明,与SARS-CoV相似,SARS-CoV-2的spike (S)蛋白包括S1蛋白和S2蛋白,两者协同完成病毒对宿主细胞的感染【3-7】。具体地,SARS-CoV-2的S蛋白通过构象改变,暴露出S1蛋白元件的RBD区域,该区域可与宿主细胞表面的ACE2蛋白结合,促进S2蛋白元件介导的病毒-宿主细胞膜融合,最终完成病毒感染【8-10】。考虑到RBD蛋白在SARS-CoV-2感染宿主细胞机制中的重要作用,针对该蛋白设计并制备中和性抗体或具有极大的前景。
基于此,2020年4月20日,陆军军医大学叶丽林团队联合重庆市公共卫生中心陈耀凯团队、中国医学科学院病原生物学研究所钱朝晖团队合作在Cellular & Molecular Immunology杂志上发表文章Human monoclonal antibodies block the binding ofSARS-CoV-2 spike protein to angiotensin convertingenzyme 2 receptor,通过功能性ELISA试验发现,近90%的COVID-19康复患者的血清中含有高水平的SARS-CoV-2 S1蛋白及RBD蛋白结合性IgG抗体。但是,研究者进一步发现,这些结合性抗体大多不能有效阻断RBD与ACE2的结合,仅有3名患者(约10%)的IgG抗体可有效地阻断RBD蛋白与ACE2蛋白的结合(图1)。

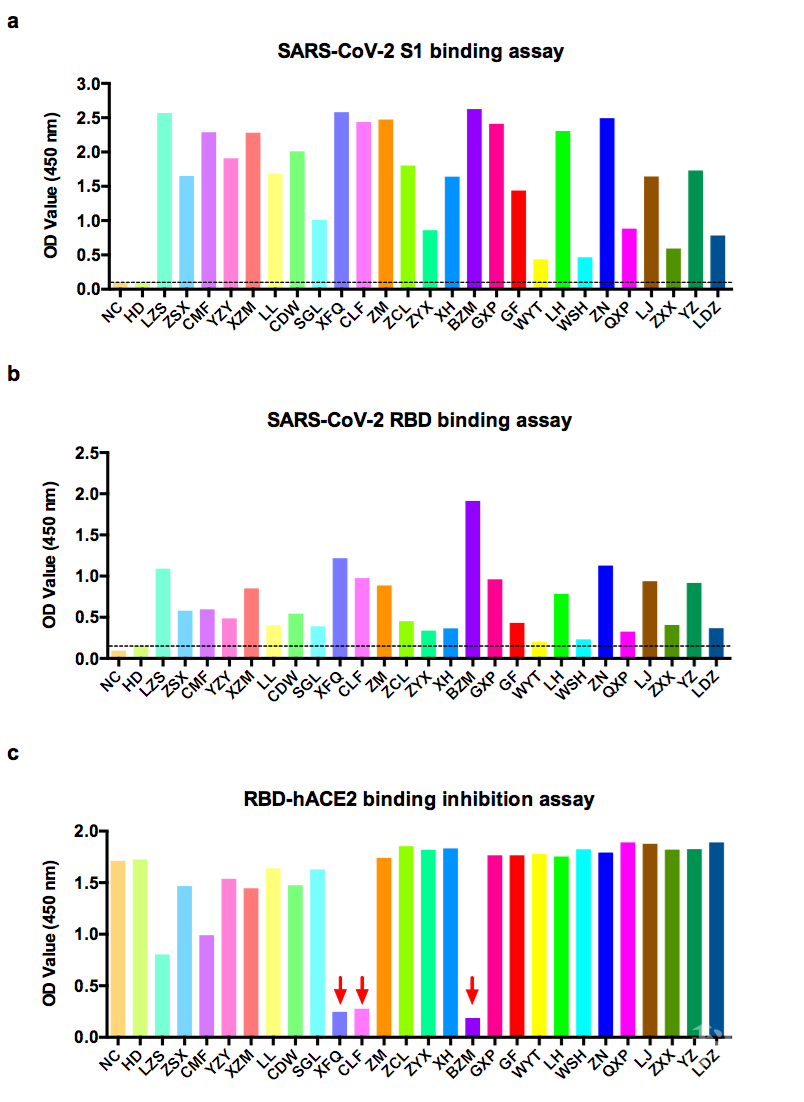 图1. COVID-19康复患者血清SARS-CoV-2 S蛋白及RBD蛋白水平及功能
图1. COVID-19康复患者血清SARS-CoV-2 S蛋白及RBD蛋白水平及功能接着,针对上述3名患者(血清SARS-CoV-2 RBD蛋白IgG抗体具有阻断RBD蛋白-ACE2蛋白结合阻断活性),研究者从其外周血中分选获得特异性识别SARS-CoV-2 RBD蛋白的记忆性B细胞,其含有编码结合RBD蛋白特异性的抗体基因。研究者进一步结合成熟的单细胞水平抗体制备技术【11】,制备并筛选出3株SARS-CoV-2 RBD蛋白单克隆抗体,包括311mab-31B5、311mab-32D4及311mab-31B9(图2)。
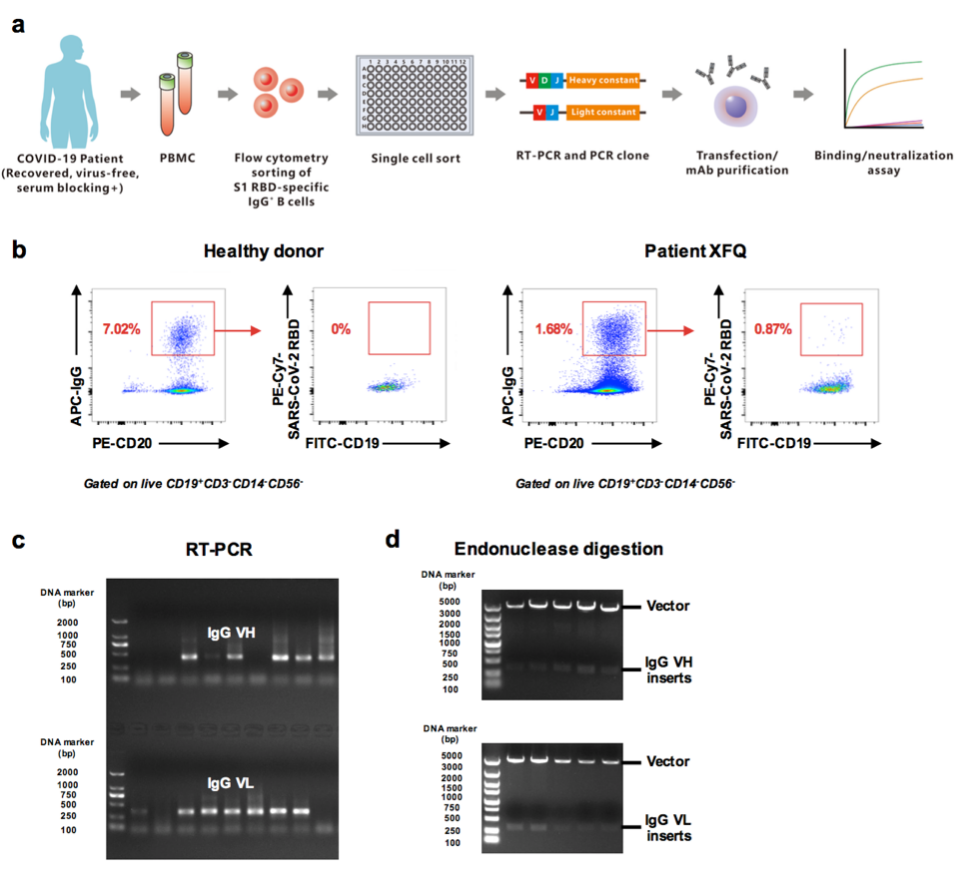 图2. SARS-CoV-2 RBD蛋白单克隆抗体制备
图2. SARS-CoV-2 RBD蛋白单克隆抗体制备最后,研究者对其制备的3株SARS-CoV-2 RBD蛋白单克隆抗体进行了如下功能性检测(图3):一、ELISA试验表明3株SARS-CoV-2 RBD蛋白单克隆抗体均具有特异性结合RBD蛋白的能力,提示抗体制备策略具有高效性;二、基于ELISA和流式细胞术的RBD蛋白-hACE2蛋白结合抑制试验提示,311mab-31B5和311mab-32D4单克隆抗体具有中和阻断RBD蛋白与hACE2蛋白结合的能力(IC50分别为0.0332 μg/ml和0.0450 μg/ml),而311mab-31B9不具有阻断活性;三、SARS-CoV-2假病毒阻断试验进一步验证了311mab-31B5和311mab-32D4单克隆抗体的RBD蛋白-ACE2蛋白结合阻断活性(IC50分别为0.0338 μg/ml和0.0698 μg/ml)。
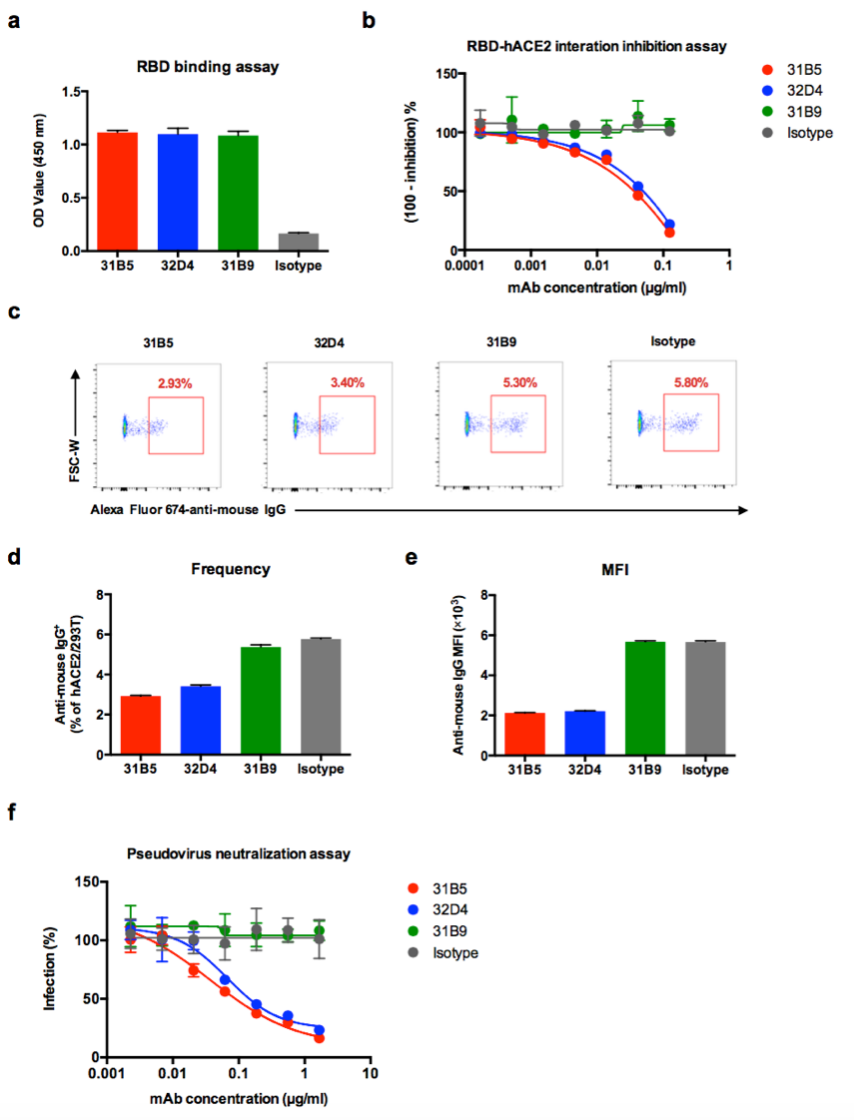 图3. SARS-CoV-2 RBD蛋白单克隆抗体的功能特性
图3. SARS-CoV-2 RBD蛋白单克隆抗体的功能特性因此,该研究中的311mab-31B5和311mab-32D4单克隆抗体可有效地阻断SARS-CoV-2 RBD蛋白与宿主ACE2蛋白的结合,具有较大的预防或治疗COVID-19的潜力。据悉,研究者目前正在测试更多的单抗克隆及其组合,并拟开展相关的体内动物试验研究。
参考文献
1. Marston, H.D., C.I. Paules, and A.S. Fauci, Monoclonal Antibodies for Emerging Infectious Diseases - Borrowing from History. N Engl J Med, 2018. 378(16): p. 1469-1472.2. Saylor, C., E. Dadachova, and A. Casadevall, Monoclonal antibody-based therapies for microbial diseases. Vaccine, 2009. 27 Suppl 6: p. G38-46.3. Zhou, P., et al., A pneumonia outbreak associated with a new coronavirus of probable bat origin. Nature, 2020. 579(7798): p. 270-273.4. Jun Lan, J.G., Jinfang Yu, Sisi Shan, Huan Zhou, Shilong Fan, Qi Zhang, Xuanling Shi, Qisheng Wang, Linqi Zhang, Xinquan Wang, Crystal structure of the 2019-nCoV spike receptor-binding domain bound with the ACE2 receptor. bioRxiv, 2020. 2020.02.19.956235; doi: https://doi.org/10.1101/2020.02.19.956235 .5. Li, F., Structure, Function, and Evolution of Coronavirus Spike Proteins. Annu Rev Virol, 2016. 3(1): p. 237-261.6. Li, F., et al., Structure of SARS coronavirus spike receptor-binding domain complexed with receptor. Science, 2005. 309(5742): p. 1864-8.7. Yan, R., et al., Structural basis for the recognition of the SARS-CoV-2 by full-length human ACE2. Science, 2020.8. Gui, M., et al., Cryo-electron microscopy structures of the SARS-CoV spike glycoprotein reveal a prerequisite conformational state for receptor binding. Cell Res, 2017. 27(1): p. 119-129.9. Kirchdoerfer, R.N., et al., Pre-fusion structure of a human coronavirus spike protein. Nature, 2016. 531(7592): p. 118-21.10. Wrapp, D., et al., Cryo-EM structure of the 2019-nCoV spike in the prefusion conformation. Science, 2020. 367(6483): p. 1260-1263.11. Smith, K., et al., Rapid generation of fully human monoclonal antibodies specific to a vaccinating antigen. Nat Protoc, 2009. 4(3): p. 372-84.
来源:BioGossip BioArt
原文链接:https://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652483565&idx=5&sn=074e6a428daecc088ca9843c36bd3817&chksm=84e23c59b395b54fbed280dd34cc072628bee841d3834b73e5604147ec6b1adcf3ce4053d86e#rd
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn



